DAY 18 推断聚类后簇的类型
@浙大疏锦行聚类后的分析:推断簇的类型
知识点回顾:
1.推断簇含义的2个思路:先选特征和后选特征
2.通过可视化图形借助ai定义簇的含义
3.科研逻辑闭环:通过精度判断特征工程价值
作业:参考示例代码对心脏病数据集采取类似操作,并且评估特征工程后模型效果有无提升。
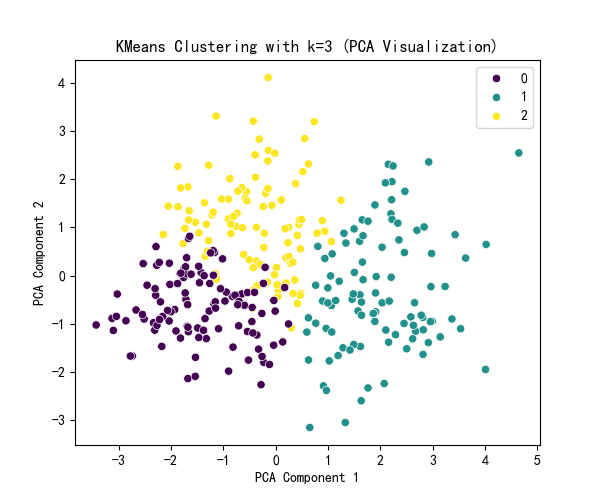
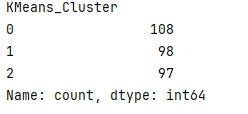
# 先运行之前预处理好的代码import pandas as pdimport pandas as pd # 用于数据处理和分析,可处理表格数据。import numpy as np # 用于数值计算,提供了高效的数组操作。import matplotlib.pyplot as plt # 用于绘制各种类型的图表import seaborn as sns # 基于matplotlib的高级绘图库,能绘制更美观的统计图形。import warningswarnings.filterwarnings(\"ignore\")# 设置中文字体(解决中文显示问题)plt.rcParams[\'font.sans-serif\'] = [\'SimHei\'] # Windows系统常用黑体字体plt.rcParams[\'axes.unicode_minus\'] = False # 正常显示负号data = pd.read_csv(\'data.csv\') # 读取数据# 先筛选字符串变量discrete_features = data.select_dtypes(include=[\'object\']).columns.tolist()# Home Ownership 标签编码home_ownership_mapping = { \'Own Home\': 1, \'Rent\': 2, \'Have Mortgage\': 3, \'Home Mortgage\': 4}data[\'Home Ownership\'] = data[\'Home Ownership\'].map(home_ownership_mapping)# Years in current job 标签编码years_in_job_mapping = { \'< 1 year\': 1, \'1 year\': 2, \'2 years\': 3, \'3 years\': 4, \'4 years\': 5, \'5 years\': 6, \'6 years\': 7, \'7 years\': 8, \'8 years\': 9, \'9 years\': 10, \'10+ years\': 11}data[\'Years in current job\'] = data[\'Years in current job\'].map(years_in_job_mapping)# Purpose 独热编码,记得需要将bool类型转换为数值data = pd.get_dummies(data, columns=[\'Purpose\'])data2 = pd.read_csv(\"data.csv\") # 重新读取数据,用来做列名对比list_final = [] # 新建一个空列表,用于存放独热编码后新增的特征名for i in data.columns: if i not in data2.columns: list_final.append(i) # 这里打印出来的就是独热编码后的特征名for i in list_final: data[i] = data[i].astype(int) # 这里的i就是独热编码后的特征名# Term 0 - 1 映射term_mapping = { \'Short Term\': 0, \'Long Term\': 1}data[\'Term\'] = data[\'Term\'].map(term_mapping)data.rename(columns={\'Term\': \'Long Term\'}, inplace=True) # 重命名列continuous_features = data.select_dtypes(include=[\'int64\', \'float64\']).columns.tolist() # 把筛选出来的列名转换成列表# 连续特征用中位数补全for feature in continuous_features: mode_value = data[feature].mode()[0] # 获取该列的众数。 data[feature].fillna(mode_value, inplace=True) # 用众数填充该列的缺失值,inplace=True表示直接在原数据上修改。# 最开始也说了 很多调参函数自带交叉验证,甚至是必选的参数,你如果想要不交叉反而实现起来会麻烦很多# 所以这里我们还是只划分一次数据集from sklearn.model_selection import train_test_splitX = data.drop([\'Credit Default\'], axis=1) # 特征,axis=1表示按列删除y = data[\'Credit Default\'] # 标签# # 按照8:2划分训练集和测试集# X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42) # 80%训练集,20%测试集import numpy as npimport pandas as pdfrom sklearn.cluster import KMeans, DBSCAN, AgglomerativeClusteringfrom sklearn.preprocessing import StandardScalerfrom sklearn.decomposition import PCAimport matplotlib.pyplot as pltimport seaborn as sns# 标准化数据(聚类前通常需要标准化)scaler = StandardScaler()X_scaled = scaler.fit_transform(X)# X_scaledimport numpy as npimport pandas as pdfrom sklearn.cluster import KMeansfrom sklearn.preprocessing import StandardScalerfrom sklearn.decomposition import PCAfrom sklearn.metrics import silhouette_score, calinski_harabasz_score, davies_bouldin_scoreimport matplotlib.pyplot as pltimport seaborn as sns# 评估不同 k 值下的指标k_range = range(2, 11) # 测试 k 从 2 到 10inertia_values = []silhouette_scores = []ch_scores = []db_scores = []for k in k_range: kmeans = KMeans(n_clusters=k, random_state=42) kmeans_labels = kmeans.fit_predict(X_scaled) inertia_values.append(kmeans.inertia_) # 惯性(肘部法则) silhouette = silhouette_score(X_scaled, kmeans_labels) # 轮廓系数 silhouette_scores.append(silhouette) ch = calinski_harabasz_score(X_scaled, kmeans_labels) # CH 指数 ch_scores.append(ch) db = davies_bouldin_score(X_scaled, kmeans_labels) # DB 指数 db_scores.append(db) print(f\"k={k}, 惯性: {kmeans.inertia_:.2f}, 轮廓系数: {silhouette:.3f}, CH 指数: {ch:.2f}, DB 指数: {db:.3f}\")# 提示用户选择 k 值selected_k = 3 # 这里选择3后面好分析,也可以根据图选择最佳的k值# 使用选择的 k 值进行 KMeans 聚类kmeans = KMeans(n_clusters=selected_k, random_state=42)kmeans_labels = kmeans.fit_predict(X_scaled)X[\'KMeans_Cluster\'] = kmeans_labels# 使用 PCA 降维到 2D 进行可视化pca = PCA(n_components=2)X_pca = pca.fit_transform(X_scaled)# KMeans 聚类结果可视化plt.figure(figsize=(6, 5))sns.scatterplot(x=X_pca[:, 0], y=X_pca[:, 1], hue=kmeans_labels, palette=\'viridis\')plt.title(f\'KMeans Clustering with k={selected_k} (PCA Visualization)\')plt.xlabel(\'PCA Component 1\')plt.ylabel(\'PCA Component 2\')plt.show()# 打印 KMeans 聚类标签的前几行print(f\"KMeans Cluster labels (k={selected_k}) added to X:\")print(X[[\'KMeans_Cluster\']].value_counts())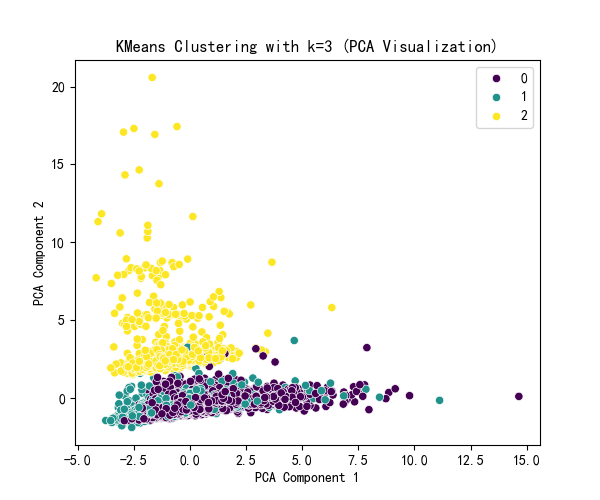

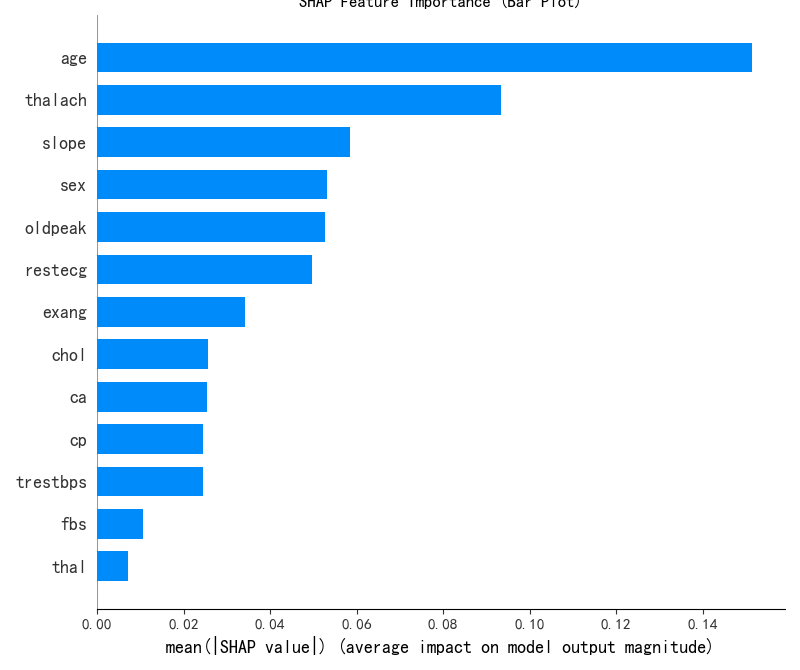
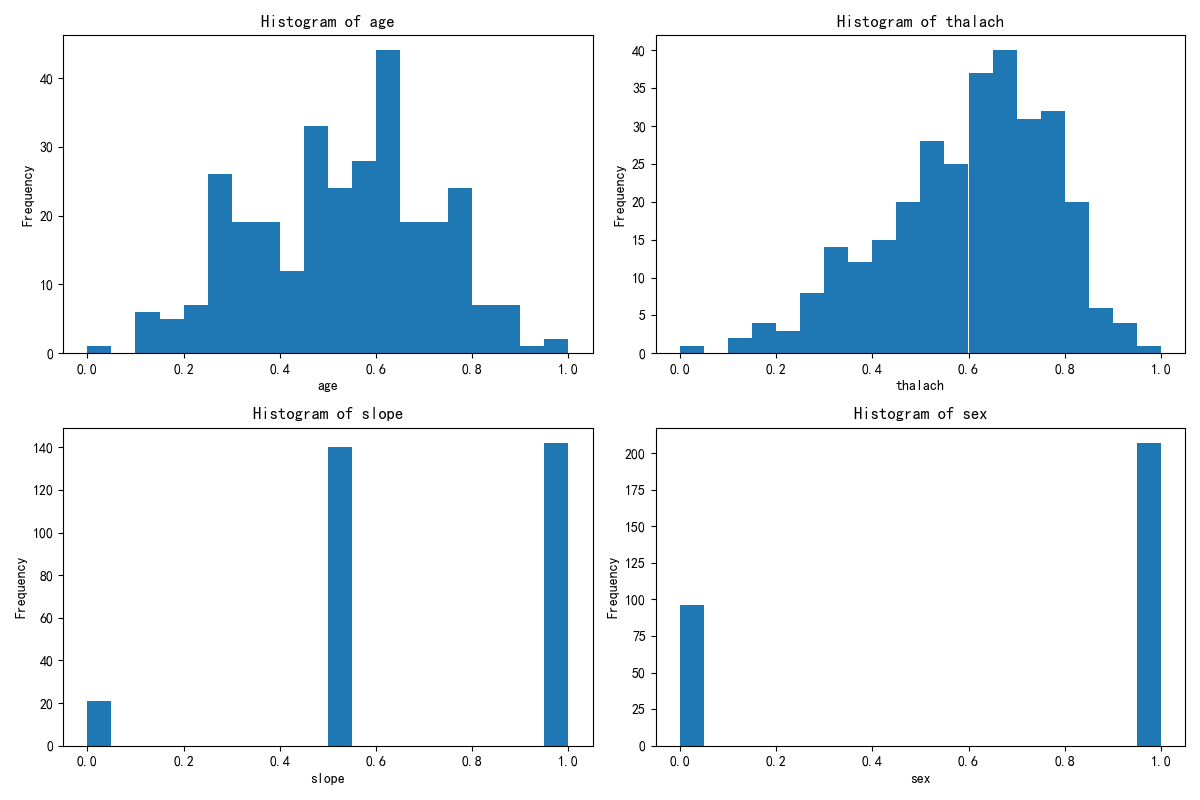
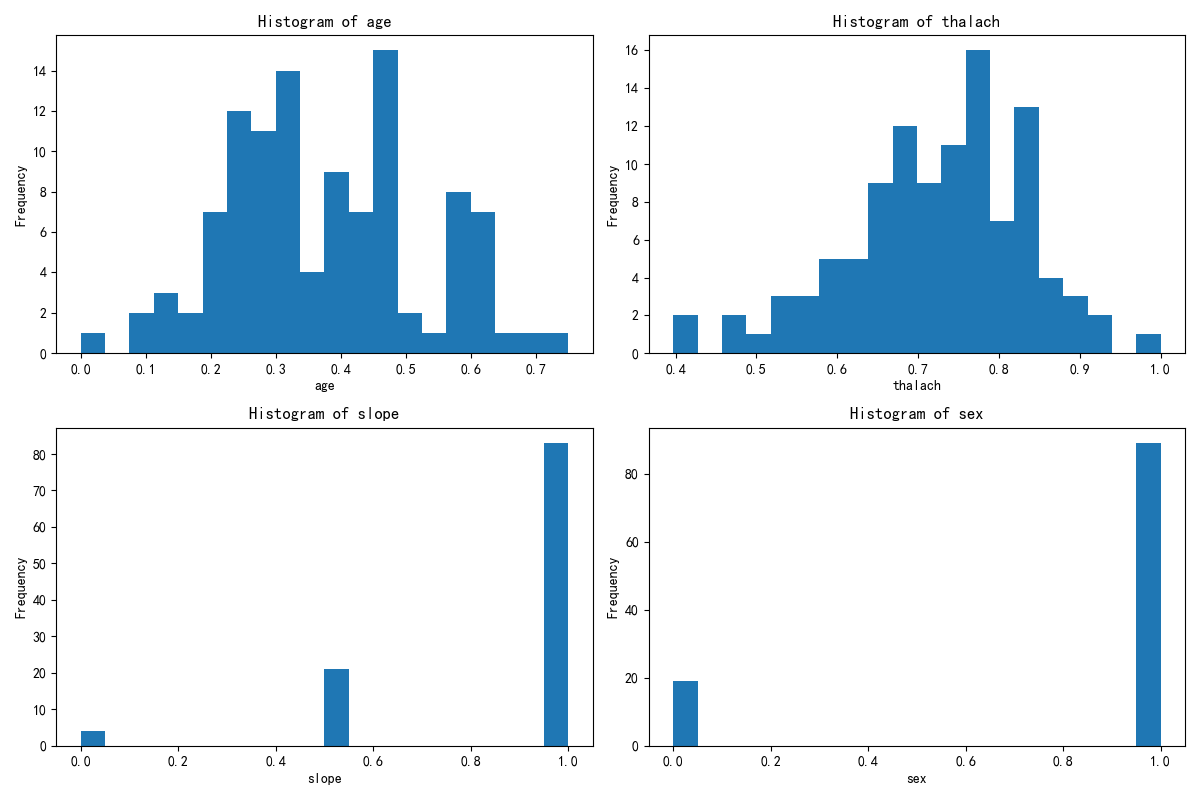
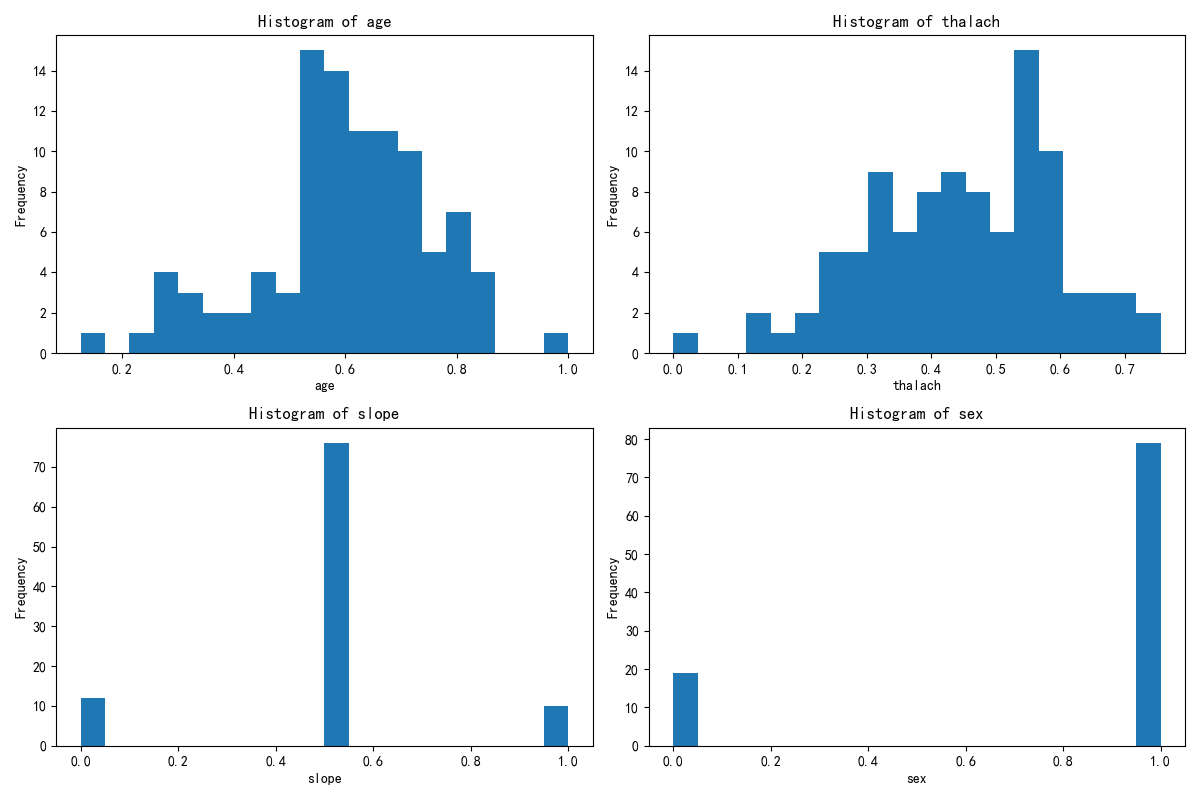
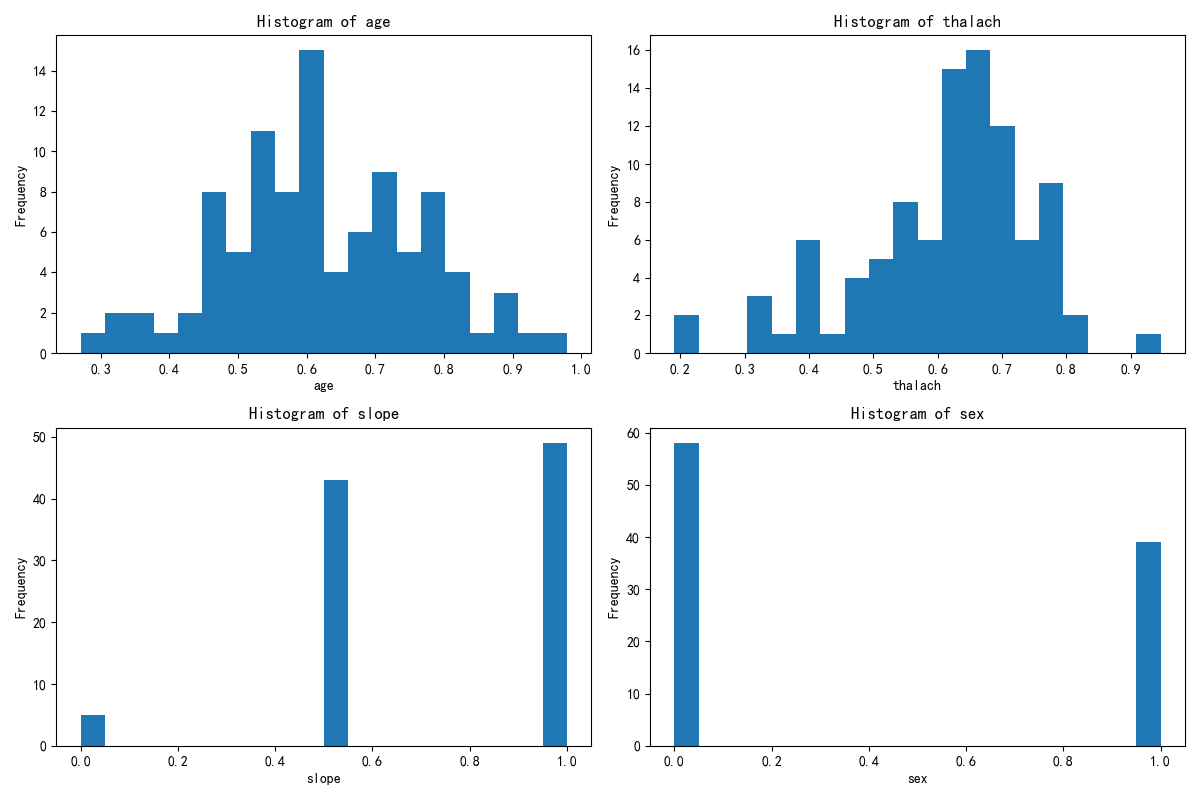
print(X.columns)x1= X.drop(\'KMeans_Cluster\',axis=1) # 删除聚类标签列y1 = X[\'KMeans_Cluster\']# 构建随机森林,用shap重要性来筛选重要性import shapimport numpy as npfrom sklearn.ensemble import RandomForestClassifier # 随机森林分类器model = RandomForestClassifier(n_estimators=100, random_state=42) # 随机森林模型model.fit(x1, y1) # 训练模型,此时无需在意准确率 直接全部数据用来训练了#通过随机森林模型和 SHAP 值来分析哪些特征对聚类结果(簇)的区分最关键#假设你已经用 K-Means 完成了聚类,给每个样本打上了 “簇标签”(存在X的KMeans_Cluster列中,比如 0、1、2 代表不同簇)。现在想知道:哪些原始特征最能解释这些簇的差异?#SHAP 是一种解释模型的工具,能量化每个特征对 “区分簇” 的影响程度。比如计算后发现 “消费金额” 的 SHAP 值最高,说明这个特征最能解释不同簇的差异。#通过这种方式,从所有原始特征中筛选出对聚类结果(簇)最关键的特征,后续就用这些特征来解释簇的含义(比如 “簇 0 的特点是消费金额高、购买频率低”)。#让模型告诉我们 “哪些特征最能区分这些簇”,从而帮我们给簇起名字、赋予含义shap.initjs()# 初始化 SHAP 解释器explainer = shap.TreeExplainer(model)shap_values = explainer.shap_values(x1) # 这个计算耗时print(shap_values.shape) # 第一维是样本数,第二维是特征数,第三维是类别数\"\"\"前面已经让 “智能识别器”(随机森林模型)学习了 “哪些苹果特点决定了它们分到 A/B/C 堆”。现在这段代码就是让这个识别器 “说人话”,告诉我们具体每个特点有多重要。shap.initjs():相当于给电脑装一个 “翻译器”,让它能把复杂的计算结果变成我们能看懂的图表(不然之后画不出图)。explainer = shap.TreeExplainer(model):给 “智能识别器” 配一个 “解说员”(SHAP 解释器),专门负责解释这个识别器是怎么判断苹果该分到哪堆的。shap_values = explainer.shap_values(x1):让 “解说员” 分析所有苹果的特点(x1),计算出 “每个特点对分堆的影响程度”,结果存在 shap_values 里。比如:“苹果大小” 这个特点,对 A 堆的影响值是 + 3(越大越可能进 A 堆)“苹果甜度” 这个特点,对 B 堆的影响值是 - 2(越甜越不可能进 B 堆)(计算起来费时间,所以代码注释说 “这个计算耗时”)shap_values.shape:查看这个 “影响程度表” 的结构。比如结果是 (100,5,3),就表示:有 100 个苹果(样本数)有 5 个分析的特点(特征数)有 3 个堆(类别数:A/B/C)简单说,这段代码就是让电脑算出 “每个苹果特点对分堆的具体影响”,为后续搞清楚 “到底是什么特点区分了不同的堆” 做准备。\"\"\"# --- 1. SHAP 特征重要性条形图 (Summary Plot - Bar) ---print(\"--- 1. SHAP 特征重要性条形图 ---\")shap.summary_plot(shap_values[:, :, 0], x1, plot_type=\"bar\",show=False) # 这里的show=False表示不直接显示图形,这样可以继续用plt来修改元素,不然就直接输出了plt.title(\"SHAP Feature Importance (Bar Plot)\")plt.show()# 此时判断一下这几个特征是离散型还是连续型import pandas as pdselected_features = [\'Purpose_debt consolidation\', \'Bankruptcies\', \'Number of Credit Problems\', \'Purpose_other\']for feature in selected_features: unique_count = X[feature].nunique() # 唯一值指的是在某一列或某个特征中,不重复出现的值 # 连续型变量通常有很多唯一值,而离散型变量的唯一值较少 print(f\'{feature} 的唯一值数量: {unique_count}\') if unique_count < 10: # 这里 10 是一个经验阈值,可以根据实际情况调整 print(f\'{feature} 可能是离散型变量\') else: print(f\'{feature} 可能是连续型变量\')\"\"\"这段代码的作用是**判断几个特定的特征(变量)是离散型还是连续型**,用的是一种简单直观的方法:通过统计每个特征中“不重复的值”(唯一值)的数量来区分。咱们用通俗的方式解释:1. 首先选了4个要分析的特征:`Purpose_debt consolidation`、`Bankruptcies`、`Number of Credit Problems`、`Purpose_other`。2. 对每个特征做两件事: - 统计它有多少个“不重复的值”(比如“性别”特征只有“男”和“女”,唯一值数量就是2)。 - 根据唯一值数量判断类型: - 如果唯一值数量少于10个(经验值),就认为是“离散型变量”(比如性别、学历这类值有限的特征)。 - 如果唯一值数量多于或等于10个,就认为是“连续型变量”(比如年龄、收入这类可以取很多不同值的特征)。3. 最后会打印出每个特征的判断结果,比如: - 假设`Bankruptcies`(破产次数)的唯一值只有0、1、2这3个,就会显示“可能是离散型变量”。 - 假设某个特征有20个不同的值,就会显示“可能是连续型变量”。简单说,这段代码就是用“特征值的丰富程度”来快速区分变量类型,帮我们更好地理解数据——因为离散型和连续型变量在后续分析(比如建模、可视化)中的处理方式是不同的。\"\"\"# X[\"Purpose_debt consolidation\"].value_counts() # 统计每个唯一值的出现次数import matplotlib.pyplot as plt# 总样本中的前四个重要性的特征分布图fig, axes = plt.subplots(2, 2, figsize=(12, 8))axes = axes.flatten()for i, feature in enumerate(selected_features): axes[i].hist(X[feature], bins=20) axes[i].set_title(f\'Histogram of {feature}\') axes[i].set_xlabel(feature) axes[i].set_ylabel(\'Frequency\')plt.tight_layout()plt.show()# 绘制出每个簇对应的这四个特征的分布图X[[\'KMeans_Cluster\']].value_counts()# 分别筛选出每个簇的数据X_cluster0 = X[X[\'KMeans_Cluster\'] == 0]X_cluster1 = X[X[\'KMeans_Cluster\'] == 1]X_cluster2 = X[X[\'KMeans_Cluster\'] == 2]\"\"\"X_cluster0 = X[X[\'KMeans_Cluster\'] == 0]:从原始数据X中,筛选出所有 “簇标签为 0” 的样本,单独存到X_cluster0里(这就是第一簇的所有数据)。X_cluster1 = X[X[\'KMeans_Cluster\'] == 1]:同理,筛选出 “簇标签为 1” 的样本,存到X_cluster1里(第二簇的数据)。X_cluster2 = X[X[\'KMeans_Cluster\'] == 2]:筛选出 “簇标签为 2” 的样本,存到X_cluster2里(第三簇的数据)。\"\"\"# 先绘制簇0的分布图import matplotlib.pyplot as plt# 总样本中的前四个重要性的特征分布图fig, axes = plt.subplots(2, 2, figsize=(12, 8))axes = axes.flatten()for i, feature in enumerate(selected_features): axes[i].hist(X_cluster0[feature], bins=20) axes[i].set_title(f\'Histogram of {feature}\') axes[i].set_xlabel(feature) axes[i].set_ylabel(\'Frequency\')plt.tight_layout()plt.show()# 绘制簇1的分布图import matplotlib.pyplot as plt# 总样本中的前四个重要性的特征分布图fig, axes = plt.subplots(2, 2, figsize=(12, 8))axes = axes.flatten()for i, feature in enumerate(selected_features): axes[i].hist(X_cluster1[feature], bins=20) axes[i].set_title(f\'Histogram of {feature}\') axes[i].set_xlabel(feature) axes[i].set_ylabel(\'Frequency\')plt.tight_layout()plt.show()# 绘制簇3的分布图import matplotlib.pyplot as plt# 总样本中的前四个重要性的特征分布图fig, axes = plt.subplots(2, 2, figsize=(12, 8))axes = axes.flatten()for i, feature in enumerate(selected_features): axes[i].hist(X_cluster2[feature], bins=20) axes[i].set_title(f\'Histogram of {feature}\') axes[i].set_xlabel(feature) axes[i].set_ylabel(\'Frequency\')plt.tight_layout()plt.show()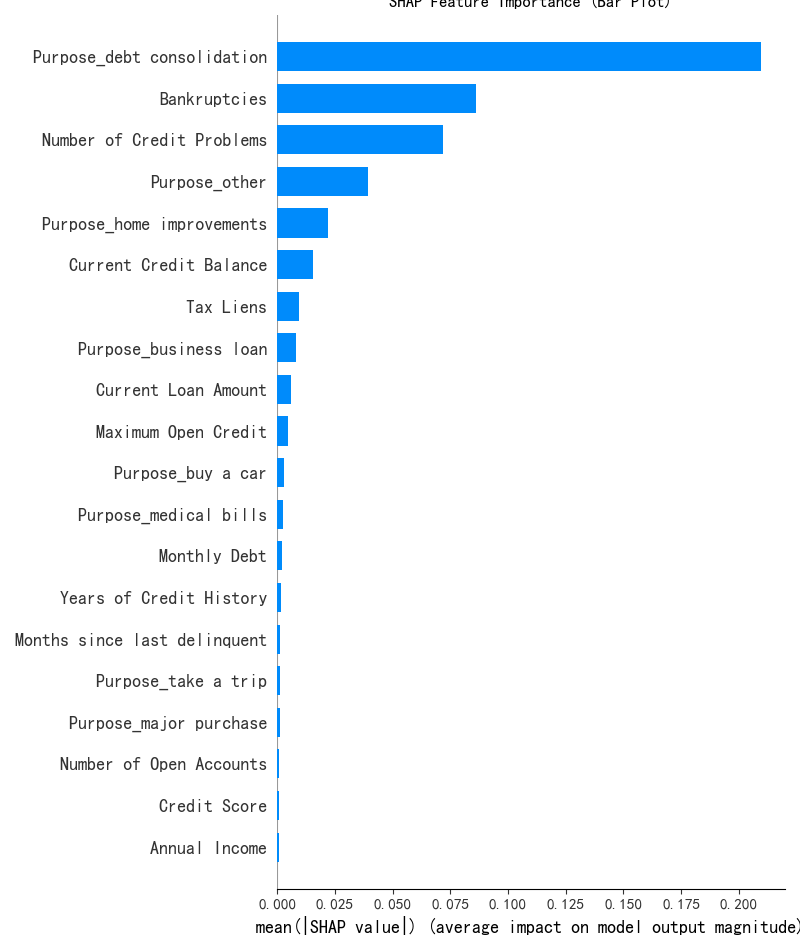
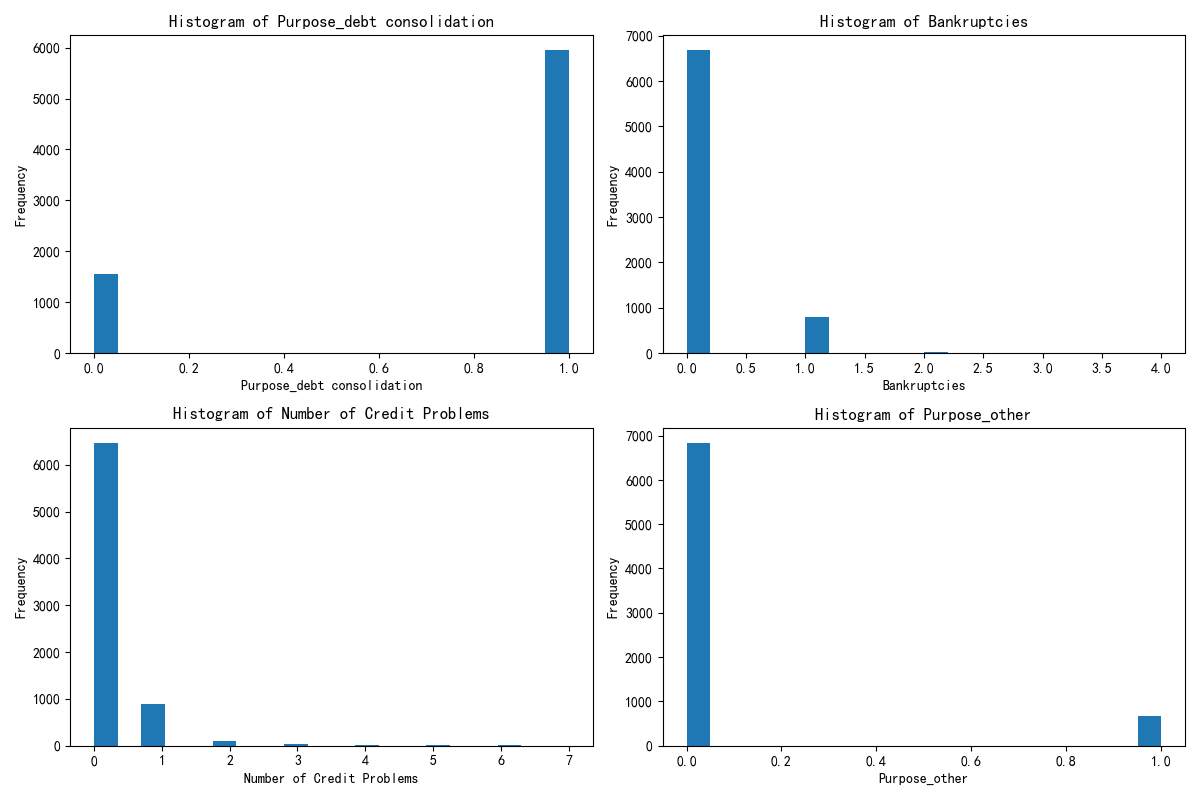
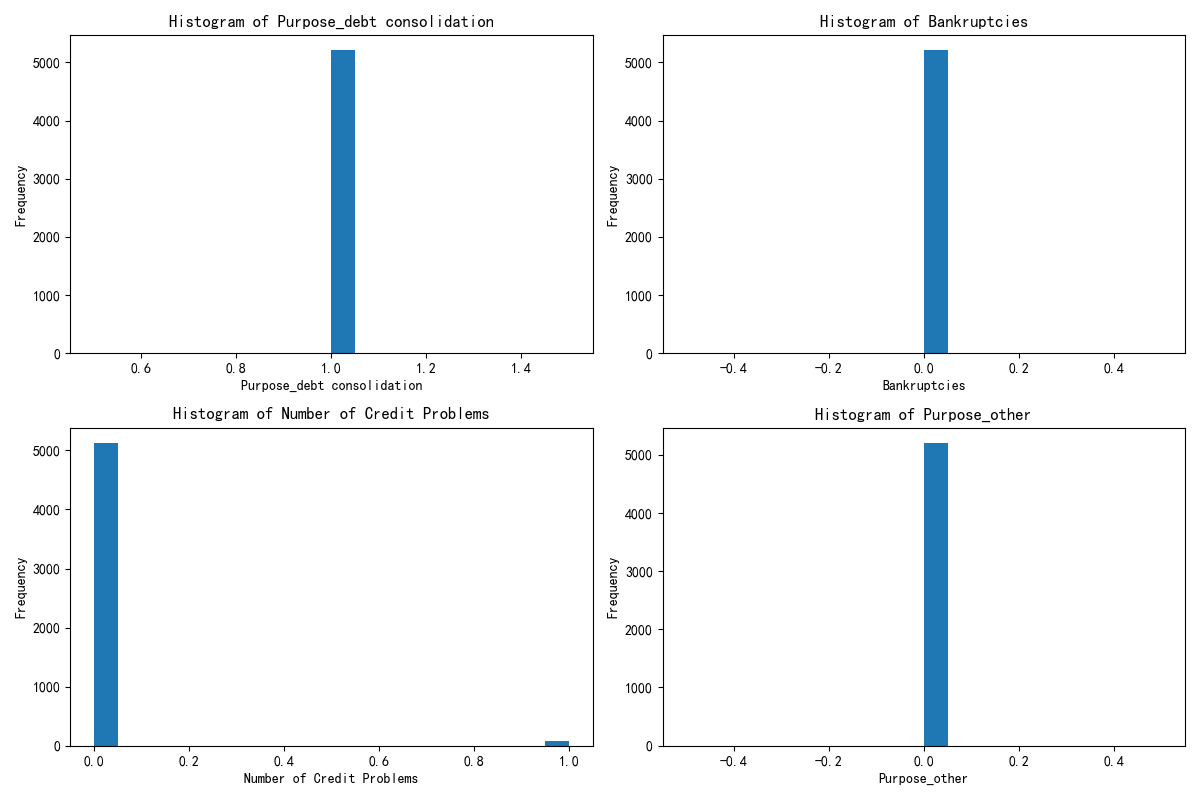
作业
#作业import pandas as pdimport pandas as pd # 用于数据处理和分析,可处理表格数据。import numpy as np # 用于数值计算,提供了高效的数组操作。import matplotlib.pyplot as plt # 用于绘制各种类型的图表import seaborn as sns # 基于matplotlib的高级绘图库,能绘制更美观的统计图形。import warningsfrom sklearn.preprocessing import MinMaxScalerwarnings.filterwarnings(\"ignore\")# 显示所有列(解决列显示不全)pd.set_option(\'display.max_columns\', None)# 显示所有行(如果数据行数不多,可开启;行数太多建议按需设置,比如 500)pd.set_option(\'display.max_rows\', None)# 扩大每列的显示宽度(避免内容被截断)pd.set_option(\'display.max_colwidth\', 100)# 设置中文字体(解决中文显示问题)plt.rcParams[\'font.sans-serif\'] = [\'SimHei\'] # Windows系统常用黑体字体plt.rcParams[\'axes.unicode_minus\'] = False # 正常显示负号data = pd.read_csv(\'heart.csv\') # 读取数据# 先筛选字符串变量discrete_features = data.select_dtypes(include=[\'object\']).columns.tolist()print(discrete_features)continuous_features = []for i in data.columns: if data[i].dtype != \'object\': continuous_features.append(i)print(continuous_features)missing_values = data.isnull().sum()from sklearn.ensemble import RandomForestClassifier #随机森林分类器from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_score # 用于评估分类器性能的指标from sklearn.metrics import classification_report, confusion_matrix #用于生成分类报告和混淆矩阵import warnings #用于忽略警告信息warnings.filterwarnings(\"ignore\") # 忽略所有警告信息from sklearn.model_selection import train_test_splitfeatures = [\'age\', \'sex\', \'cp\', \'trestbps\', \'chol\', \'fbs\', \'restecg\', \'thalach\', \'exang\', \'oldpeak\', \'slope\', \'ca\', \'thal\', \'target\']# 初始化归一化器scaler = MinMaxScaler()# 对选定列进行归一化data[features] = scaler.fit_transform(data[features])# 检查归一化后的数据print(data.head())from sklearn.model_selection import train_test_splitX = data.drop([\'target\'], axis=1) # 特征,axis=1表示按列删除y = data[\'target\'] # 标签# # 按照8:2划分训练集和测试集# X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42) # 80%训练集,20%测试集import numpy as npimport pandas as pdfrom sklearn.cluster import KMeans, DBSCAN, AgglomerativeClusteringfrom sklearn.preprocessing import StandardScalerfrom sklearn.decomposition import PCAimport matplotlib.pyplot as pltimport seaborn as sns#KMeans 是基于距离的算法,如果特征的尺度差异大(比如一个特征是 “年龄”(0-100),另一个是 “收入”(0-10000)),距离会被大尺度特征主导,导致聚类结果失真。#StandardScaler会把所有特征转换为 “均值 0、标准差 1” 的分布,让每个特征对距离的影响平等。# 标准化数据(聚类前通常需要标准化)scaler = StandardScaler()X_scaled = scaler.fit_transform(X)# X_scaledimport numpy as npimport pandas as pdfrom sklearn.cluster import KMeansfrom sklearn.preprocessing import StandardScalerfrom sklearn.decomposition import PCAfrom sklearn.metrics import silhouette_score, calinski_harabasz_score, davies_bouldin_scoreimport matplotlib.pyplot as pltimport seaborn as sns# 评估不同 k 值下的指标k_range = range(2, 11) # 测试 k 从 2 到 10#作用:测试聚类数k从2到10,因为k=1无意义。inertia_values = []silhouette_scores = []ch_scores = []db_scores = []#作用:用于存储每个k值对应的评估结果,后续绘图用。for k in k_range: kmeans = KMeans(n_clusters=k, random_state=42) kmeans_labels = kmeans.fit_predict(X_scaled) inertia_values.append(kmeans.inertia_) # 惯性(肘部法则) silhouette = silhouette_score(X_scaled, kmeans_labels) # 轮廓系数 silhouette_scores.append(silhouette) ch = calinski_harabasz_score(X_scaled, kmeans_labels) # CH 指数 ch_scores.append(ch) db = davies_bouldin_score(X_scaled, kmeans_labels) # DB 指数 db_scores.append(db) print(f\"k={k}, 惯性: {kmeans.inertia_:.2f}, 轮廓系数: {silhouette:.3f}, CH 指数: {ch:.2f}, DB 指数: {db:.3f}\")# 提示用户选择 k 值selected_k = 3# 使用选择的 k 值进行 KMeans 聚类kmeans = KMeans(n_clusters=selected_k, random_state=42)kmeans_labels = kmeans.fit_predict(X_scaled)X[\'KMeans_Cluster\'] = kmeans_labels# 使用 PCA 降维到 2D 进行可视化pca = PCA(n_components=2)X_pca = pca.fit_transform(X_scaled)# KMeans 聚类结果可视化plt.figure(figsize=(6, 5))sns.scatterplot(x=X_pca[:, 0], y=X_pca[:, 1], hue=kmeans_labels, palette=\'viridis\')plt.title(f\'KMeans Clustering with k={selected_k} (PCA Visualization)\')plt.xlabel(\'PCA Component 1\')plt.ylabel(\'PCA Component 2\')plt.show()# 打印 KMeans 聚类标签的前几行print(f\"KMeans Cluster labels (k={selected_k}) added to X:\")print(X[[\'KMeans_Cluster\']].value_counts())print(X.columns)x1= X.drop(\'KMeans_Cluster\',axis=1) # 删除聚类标签列y1 = X[\'KMeans_Cluster\']# 构建随机森林,用shap重要性来筛选重要性import shapimport numpy as npfrom sklearn.ensemble import RandomForestClassifier # 随机森林分类器model = RandomForestClassifier(n_estimators=100, random_state=42) # 随机森林模型model.fit(x1, y1) # 训练模型,此时无需在意准确率 直接全部数据用shap.initjs()# 初始化 SHAP 解释器explainer = shap.TreeExplainer(model)shap_values = explainer.shap_values(x1) # 这个计算耗时shap_values.shape # 第一维是样本数,第二维是特征数,第三维是类别数# --- 1. SHAP 特征重要性条形图 (Summary Plot - Bar) ---print(\"--- 1. SHAP 特征重要性条形图 ---\")shap.summary_plot(shap_values[:, :, 0], x1, plot_type=\"bar\",show=False) # 这里的show=False表示不直接显示图形,这样可以继续用plt来修改元素,不然就直接输出了plt.title(\"SHAP Feature Importance (Bar Plot)\")plt.show()# 此时判断一下这几个特征是离散型还是连续型import pandas as pdselected_features = [\'age\',\'thalach\',\'slope\',\'sex\']for feature in selected_features: unique_count = X[feature].nunique() # 唯一值指的是在某一列或某个特征中,不重复出现的值 # 连续型变量通常有很多唯一值,而离散型变量的唯一值较少 print(f\'{feature} 的唯一值数量: {unique_count}\') if unique_count < 10: # 这里 10 是一个经验阈值,可以根据实际情况调整 print(f\'{feature} 可能是离散型变量\') else: print(f\'{feature} 可能是连续型变量\')# X[\"Purpose_debt consolidation\"].value_counts() # 统计每个唯一值的出现次数import matplotlib.pyplot as plt# 总样本中的前四个重要性的特征分布图fig, axes = plt.subplots(2, 2, figsize=(12, 8))axes = axes.flatten()for i, feature in enumerate(selected_features): axes[i].hist(X[feature], bins=20) axes[i].set_title(f\'Histogram of {feature}\') axes[i].set_xlabel(feature) axes[i].set_ylabel(\'Frequency\')plt.tight_layout()plt.show()print(X[[\'KMeans_Cluster\']].value_counts())# 分别筛选出每个簇的数据X_cluster0 = X[X[\'KMeans_Cluster\'] == 0]X_cluster1 = X[X[\'KMeans_Cluster\'] == 1]X_cluster2 = X[X[\'KMeans_Cluster\'] == 2]# 先绘制簇0的分布图import matplotlib.pyplot as plt# 总样本中的前四个重要性的特征分布图fig, axes = plt.subplots(2, 2, figsize=(12, 8))axes = axes.flatten()for i, feature in enumerate(selected_features): axes[i].hist(X_cluster0[feature], bins=20) axes[i].set_title(f\'Histogram of {feature}\') axes[i].set_xlabel(feature) axes[i].set_ylabel(\'Frequency\')plt.tight_layout()plt.show()# 绘制簇1的分布图import matplotlib.pyplot as plt# 总样本中的前四个重要性的特征分布图fig, axes = plt.subplots(2, 2, figsize=(12, 8))axes = axes.flatten()for i, feature in enumerate(selected_features): axes[i].hist(X_cluster1[feature], bins=20) axes[i].set_title(f\'Histogram of {feature}\') axes[i].set_xlabel(feature) axes[i].set_ylabel(\'Frequency\')plt.tight_layout()plt.show()# 绘制簇2的分布图import matplotlib.pyplot as plt# 总样本中的前四个重要性的特征分布图fig, axes = plt.subplots(2, 2, figsize=(12, 8))axes = axes.flatten()for i, feature in enumerate(selected_features): axes[i].hist(X_cluster2[feature], bins=20) axes[i].set_title(f\'Histogram of {feature}\') axes[i].set_xlabel(feature) axes[i].set_ylabel(\'Frequency\')plt.tight_layout()plt.show()cluster_onehot = pd.get_dummies(X[\'KMeans_Cluster\'], prefix=\'Cluster\')X_original = X.drop(\'KMeans_Cluster\', axis=1)X_new = pd.concat([X_original, cluster_onehot], axis=1)from sklearn.model_selection import train_test_splitfrom sklearn.ensemble import RandomForestClassifier # 随机森林分类器from sklearn.metrics import accuracy_score, classification_report# 1. 划分训练集和测试集(用包含目标变量y的数据)# 假设y是你要预测的标签(比如心脏病target)X_train_new, X_test_new, y_train, y_test = train_test_split(X_new, y, test_size=0.2, random_state=42)# 2. 训练“加了新特征”的随机森林模型rf_new = RandomForestClassifier(n_estimators=100, random_state=42)rf_new.fit(X_train_new, y_train) # 用包含簇特征的数据训练# 3. 预测并评估精度y_pred_new = rf_new.predict(X_test_new)new_accuracy = accuracy_score(y_test, y_pred_new)print(f\"加新特征后的精度:{new_accuracy:.4f}\")X_train_old, X_test_old, _, _ = train_test_split(X_original, y, test_size=0.2, random_state=42)rf_old = RandomForestClassifier(n_estimators=100, random_state=42)rf_old.fit(X_train_old, y_train)# 评估基准模型精度y_pred_old = rf_old.predict(X_test_old)old_accuracy = accuracy_score(y_test, y_pred_old)print(f\"不加新特征的精度:{old_accuracy:.4f}\")