从0开始学习R语言--Day57--SCAD模型
在之前,我们提到过对于基因数据,我们会倾向于用弹性网络去建模,这样可以做到节省大量计算量的同时,保留关键的变量做筛选;但是实际上弹性网络本质上是用两种方法的结合去拟合,得到的函数是凸函数,从而不可避免的会产生偏差。
相比之下,SCAD则选择更直接的分段建模,这样可以最大程度地保留原有特征的特点,同时做到更优的筛选,只是计算复杂度会更高,需要谨慎使用。
以下是一个例子:
# 加载必要的包library(ncvreg)library(ggplot2)# 生成模拟数据集set.seed(123)n <- 200p <- 10X <- matrix(rnorm(n * p), n, p)colnames(X) <- paste0(\"X\", 1:p)true_beta <- c(3, 1.5, 2, rep(0, p-3))y <- X %*% true_beta + rnorm(n, sd = 1.5)# 使用SCAD进行变量选择scad_fit <- ncvreg(X, y, penalty = \"SCAD\")# 使用交叉验证选择最优λcv_fit <- cv.ncvreg(X, y, penalty = \"SCAD\")# 查看交叉验证结果#print(cv_fit)# 获取最优λ值 (这里使用最小化误差的λ)best_lambda <- cv_fit$lambda.min# 查看最优模型的摘要summary(scad_fit, lambda = best_lambda)# 查看最优模型的系数coef(scad_fit, lambda = best_lambda)# 绘制系数路径图plot(scad_fit)abline(v = log(best_lambda), lty = 2, col = \"red\") # 标记最优λ位置# 可视化比较results <- data.frame( Variable = colnames(X), True = true_beta, SCAD = coef(scad_fit, lambda = best_lambda)[-1] # 去掉截距项)ggplot(results, aes(x = Variable)) + geom_point(aes(y = True, color = \"True\"), size = 3) + geom_point(aes(y = SCAD, color = \"SCAD\"), size = 3) + labs(title = paste(\"真实系数与SCAD估计比较 (λ =\", round(best_lambda, 4), \")\"), y = \"系数值\", color = \"类型\") + theme_minimal()输出:
SCAD-penalized linear regression with n=200, p=10At lambda=0.3861:------------------------------------------------- Nonzero coefficients : 3 Expected nonzero coefficients: 0.00 Average mfdr (3 features) : 0.000 Estimate z mfdr SelectedX1 3.003 30.39 < 1e-04 *X3 2.014 20.85 < 1e-04 *X2 1.547 16.53 < 1e-04 *(Intercept) X1 X2 X3 X4 X5 X6 X7 -0.1908806 3.0030121 1.5470496 2.0140936 0.0000000 0.0000000 0.0000000 0.0000000 X8 X9 X10 0.0000000 0.0000000 0.0000000 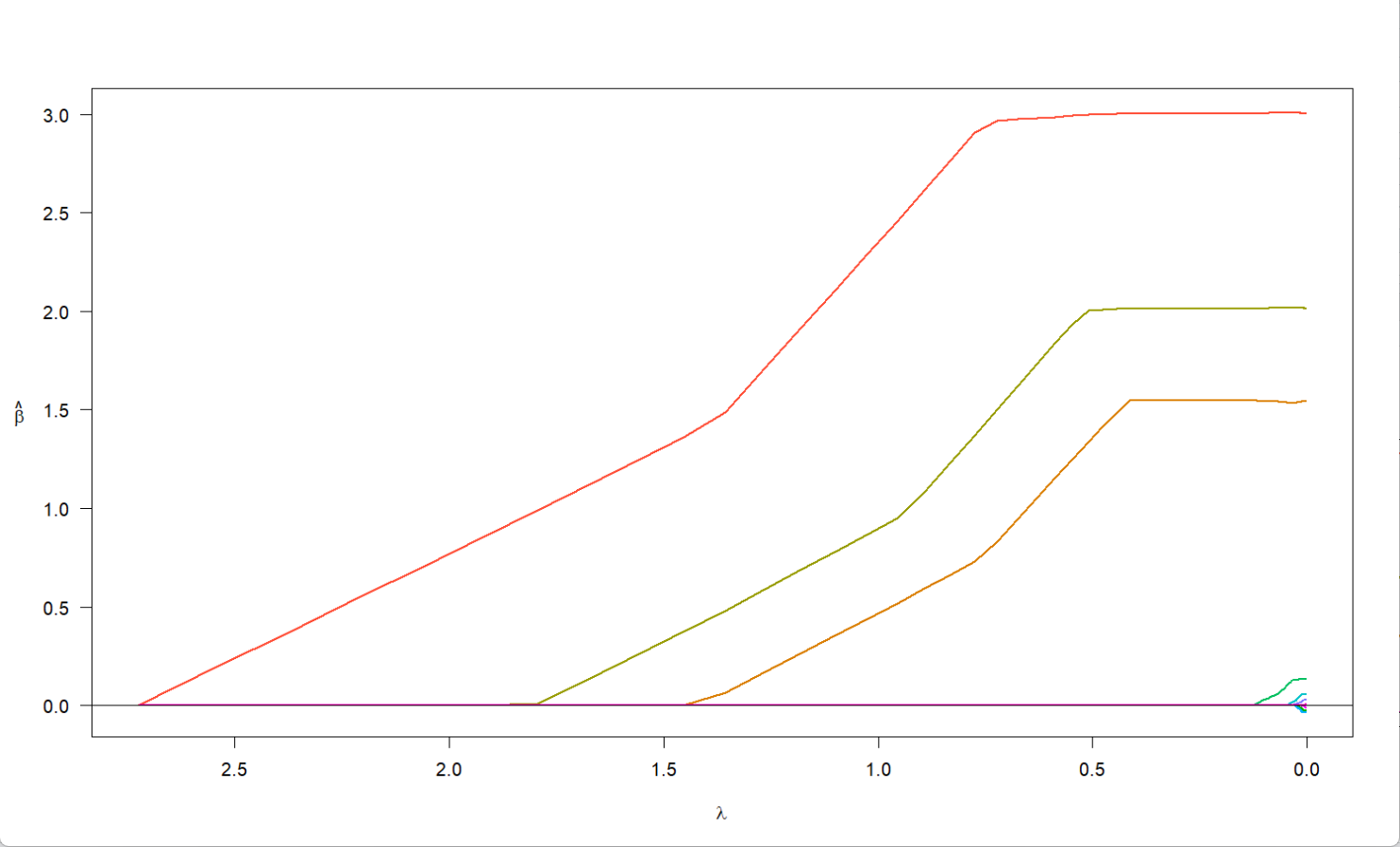
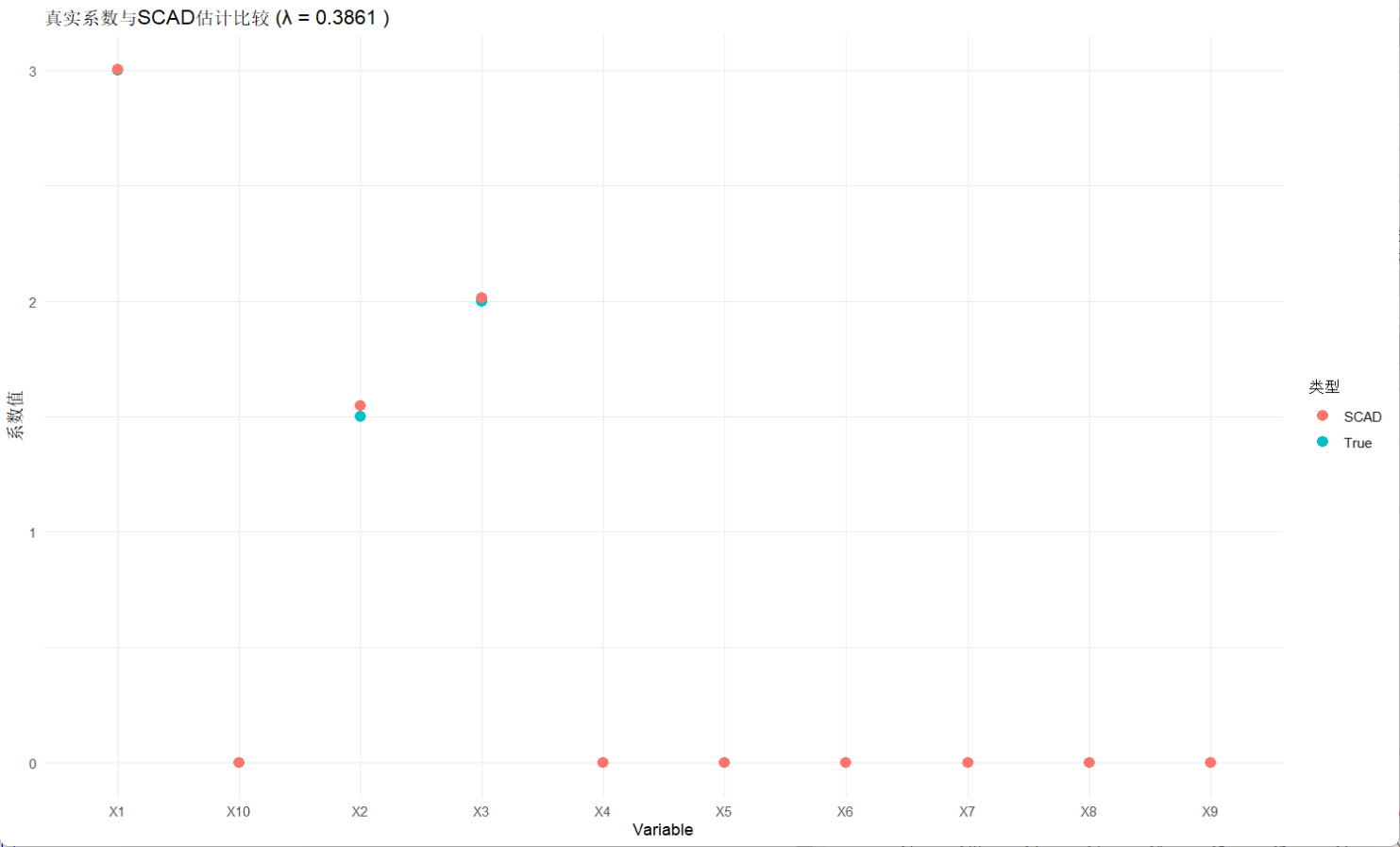
结果表明,X1、X2、X3系数非零,且mfdr < 1e-04,统计学显著,是显著变量;mdfr值极低,说明假阳性的风险极低;当lambda较大时,系数基本被压缩为0,说明模型趋于稀疏;而lambda在减少时,系数先快速上升再逐渐趋于平缓,说明其具有收敛性和稳定性。


