多组学数据库Harmonizome 3.0
摘要
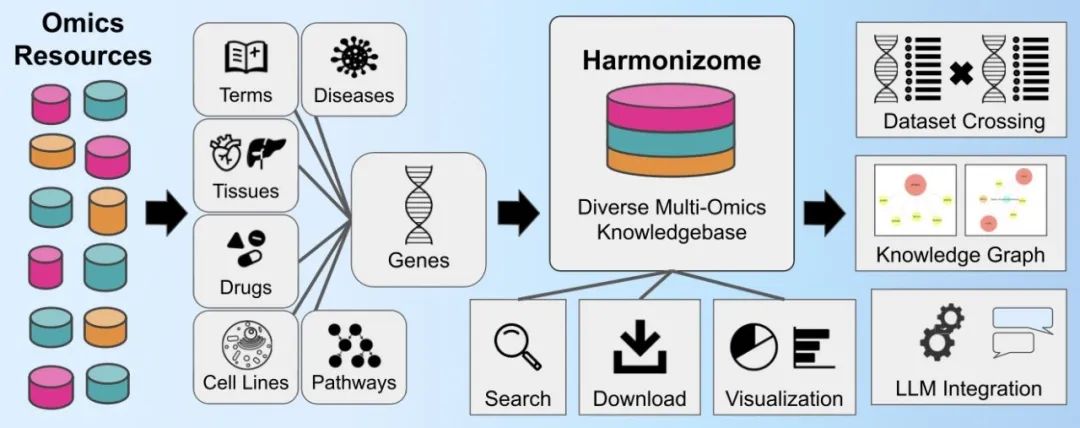
Harmonizome数据库处理和抽象多种组学数据集,将其转化为基因与其属性之间的关联,使研究人员探索和整合许多核心组学资源的人类基因知识。介绍了对原始Harmonizome数据库的重大升级Harmonizome 3.0。增加了26个数据集,提供了近1,200万条基因与各种属性类型(如细胞和组织、疾病和通路)之间的关联。还包括跨数据集的功能,识别在不同数据集中共享的基因模块。为进一步解释数据集对之间显著的高基因集合重叠,大型语言模型(LLM)会撰写一段文字,推测高重叠背后的原因。还增加了更多的数据格式和可视化选项。数据集可作为知识图谱(KG)下载,并使用统一流形逼近和投影(UMAP)图可视化。知识图谱可通过用户界面探索,将基因-属性关联可视化为球棒图。Harmonizome 3.0 是个丰富的处理过的组学数据集资源,提供多种人工智能就绪格式。
https://maayanlab.cloud/Harmonizome/
结果
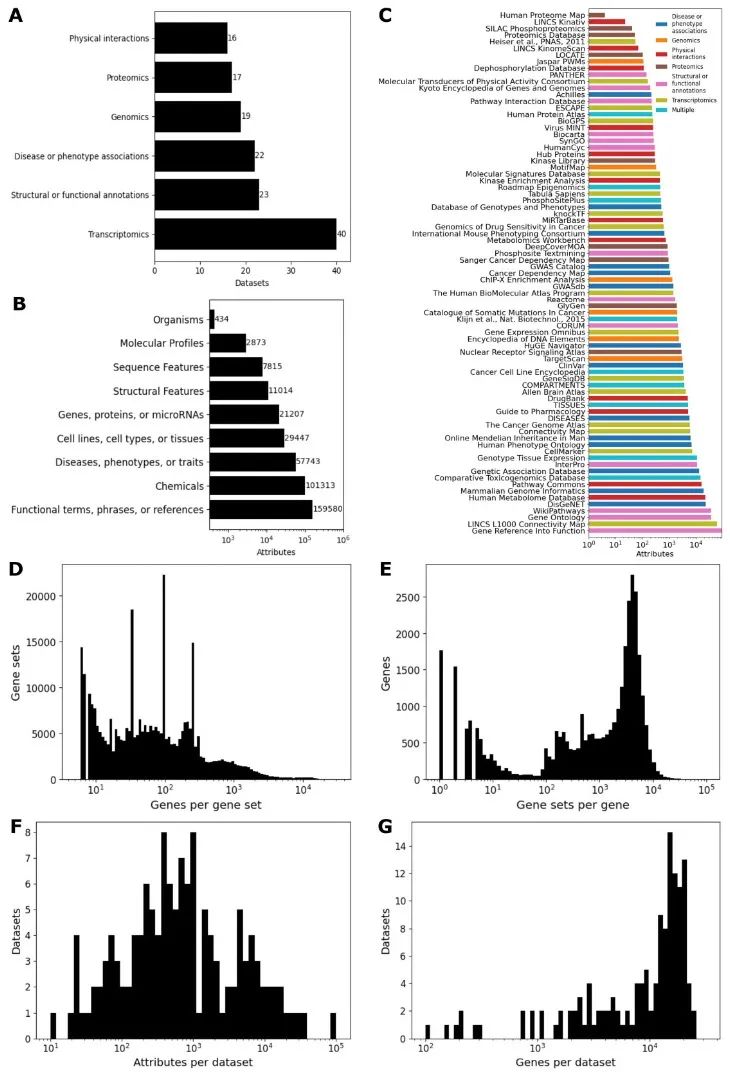
图1 Harmonizome 3.0中数据集和属性的统计摘要。 (A) 每个组所属的数据集。 (B) 每个属性组所属的基因集合。 (C) 每个资源来源的基因集合。 (D) 基因集合长度的分布。 (E) 基因在基因集合中的包含分布。 (F) 数据集属性覆盖率的分布。 (G) 数据集基因覆盖率的分布。
表1 添加到Harmonizome 3.0升级中的数据集摘要。数据集按属性组、相关出版物、属性、基因以及基因集合长度的平均值和范围列示。
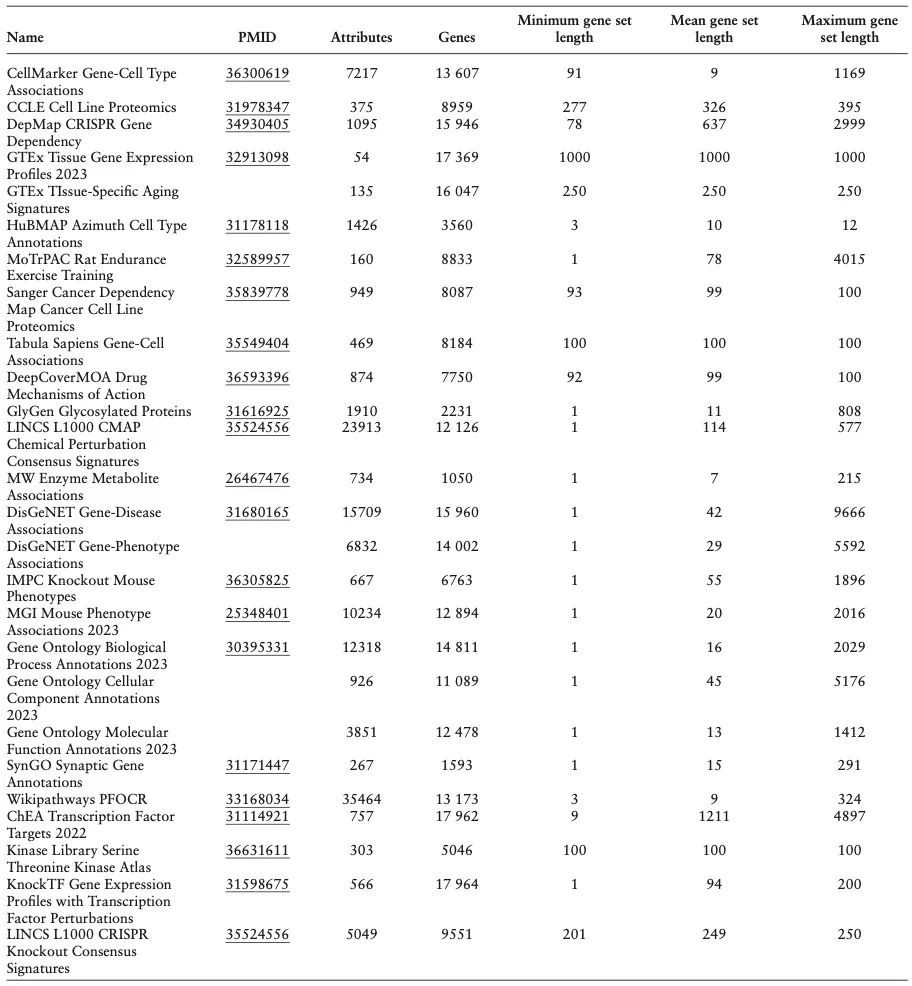
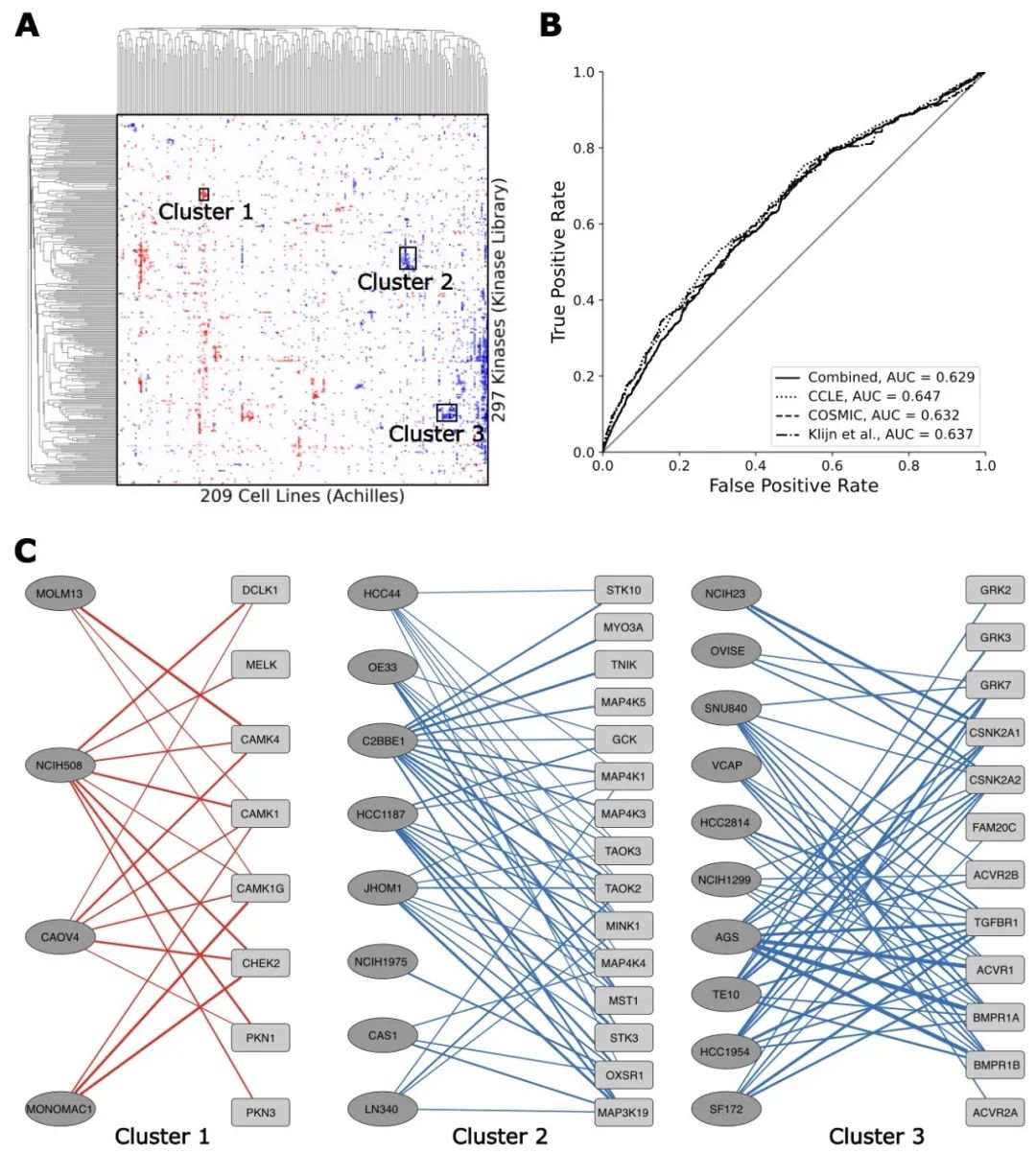
图2 将激酶库丝氨酸苏氨酸激酶组图谱数据集与Achilles细胞系基因必需性谱型数据集交叉分析。(A) 分层聚类热图显示基于其重叠显著性的激酶和细胞系之间的余弦相似性。识别出3个簇以供进一步检查。(B) 受试者工作特征(ROC)曲线评估每个交叉的重叠基因与来自Harmonizome的COSMIC、CCLE和Klijn等数据集中已知突变的一致性。(C) 每个簇中识别出的激酶和细胞系的子网络。红和蓝线条分别表示细胞系适应性的增加和减少。线条宽度与-log(P)值的大小成正比。
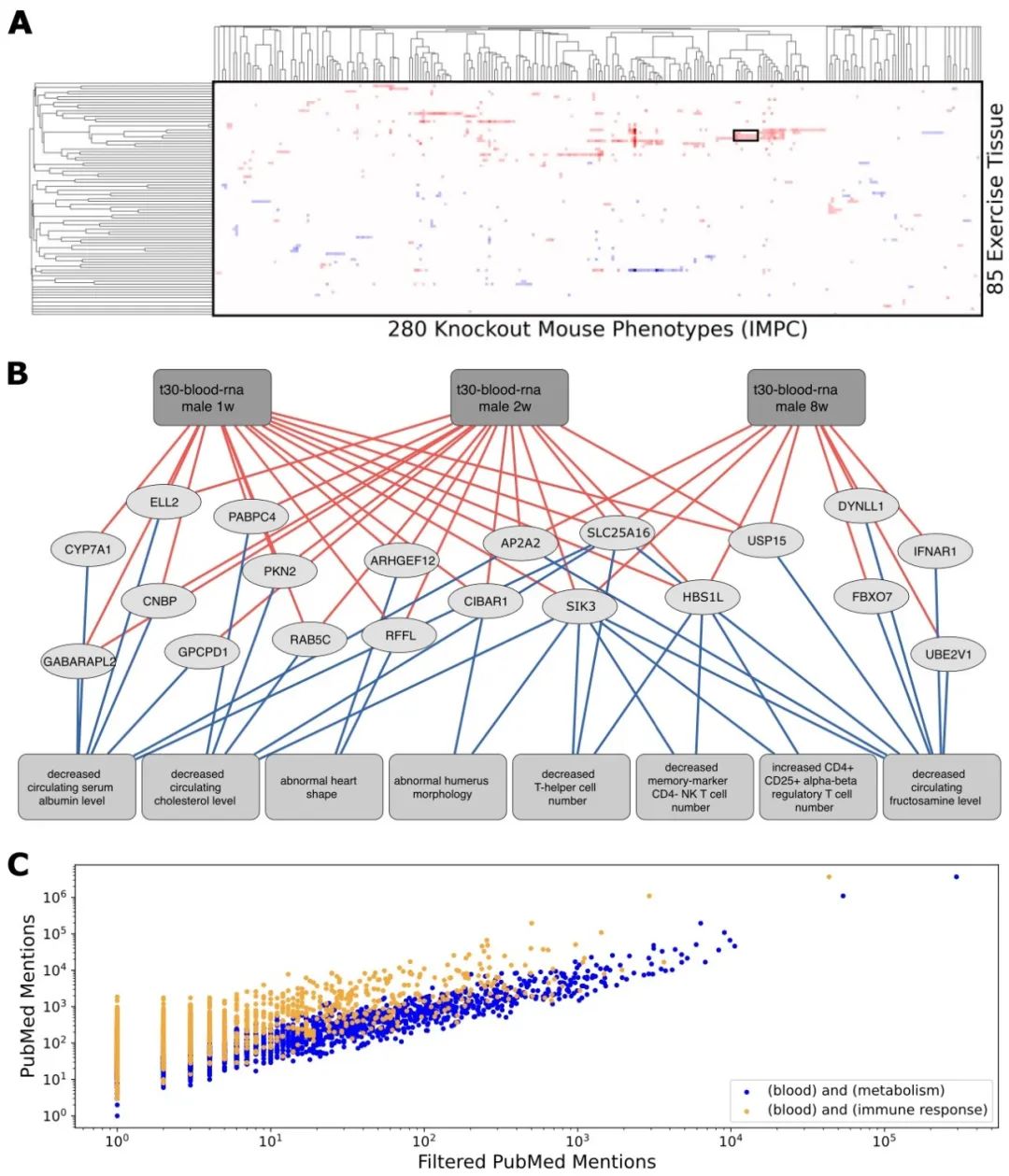
图3 将MoTrPAC大鼠耐力运动训练数据集与IMPC敲除小鼠表型数据集交叉分析。(A) 分层聚类热图显示基于重叠显著性的大鼠耐力运动训练组织样本与敲除小鼠表型之间的余弦相似性。识别出1个簇以供进一步检查。(B) 聚类组织样本、表型和重叠基因的子网络。从顶部框到基因的线条表示在耐力运动训练后表达增加的基因,到底部框的线条表示基因敲除后的表型存在。(C) 散点图比较了在运动组织样本和敲除表型之间重叠最高的所有基因在PubMed中的提及次数。PubMed提及次数是使用基因符号查询PubMed API获得的,并查询带有“血液”、“代谢”和“免疫反应”等术语的基因符号来检索提及次数,以评估已知的相关性。
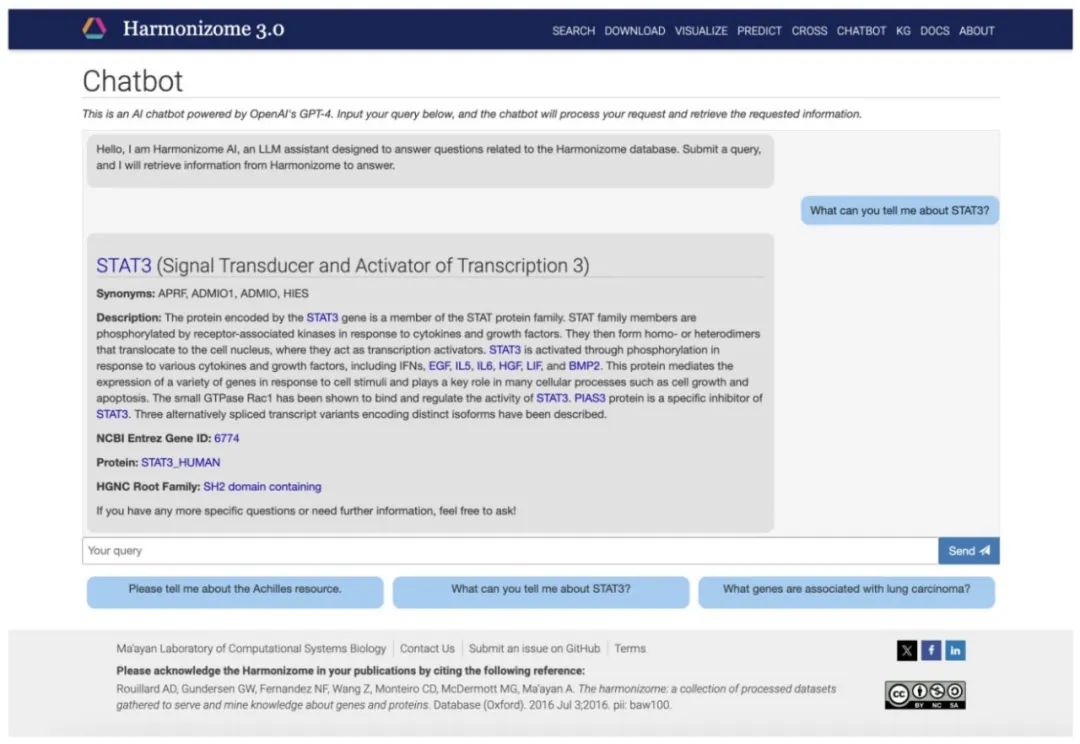
图4 Harmonizome 3.0聊天机器人的示例。选择示例查询“你能告诉我关于STAT3的哪些信息?”来向聊天机器人询问转录因子STAT3的信息。该查询通过后端Harmonizome API控制器传递给OpenAI Assistant。Assistant返回函数调用来从Harmonizome数据库中检索关于该基因的信息。控制器解析该函数调用,并将相关的元数据作为工具输出返回给Assistant。Assistant将基因的元数据重新格式化为人类可读的格式,并将其返回给API控制器。然后,控制器为用户渲染聊天历史记录,提供基因名称、功能描述、NCBI Entrez基因ID、编码蛋白和蛋白家族。在渲染的响应中,基因符号以及Harmonizome中的资源和数据集名称被超链接到相应的Harmonizome页面,以便用户进一步了解相关信息。
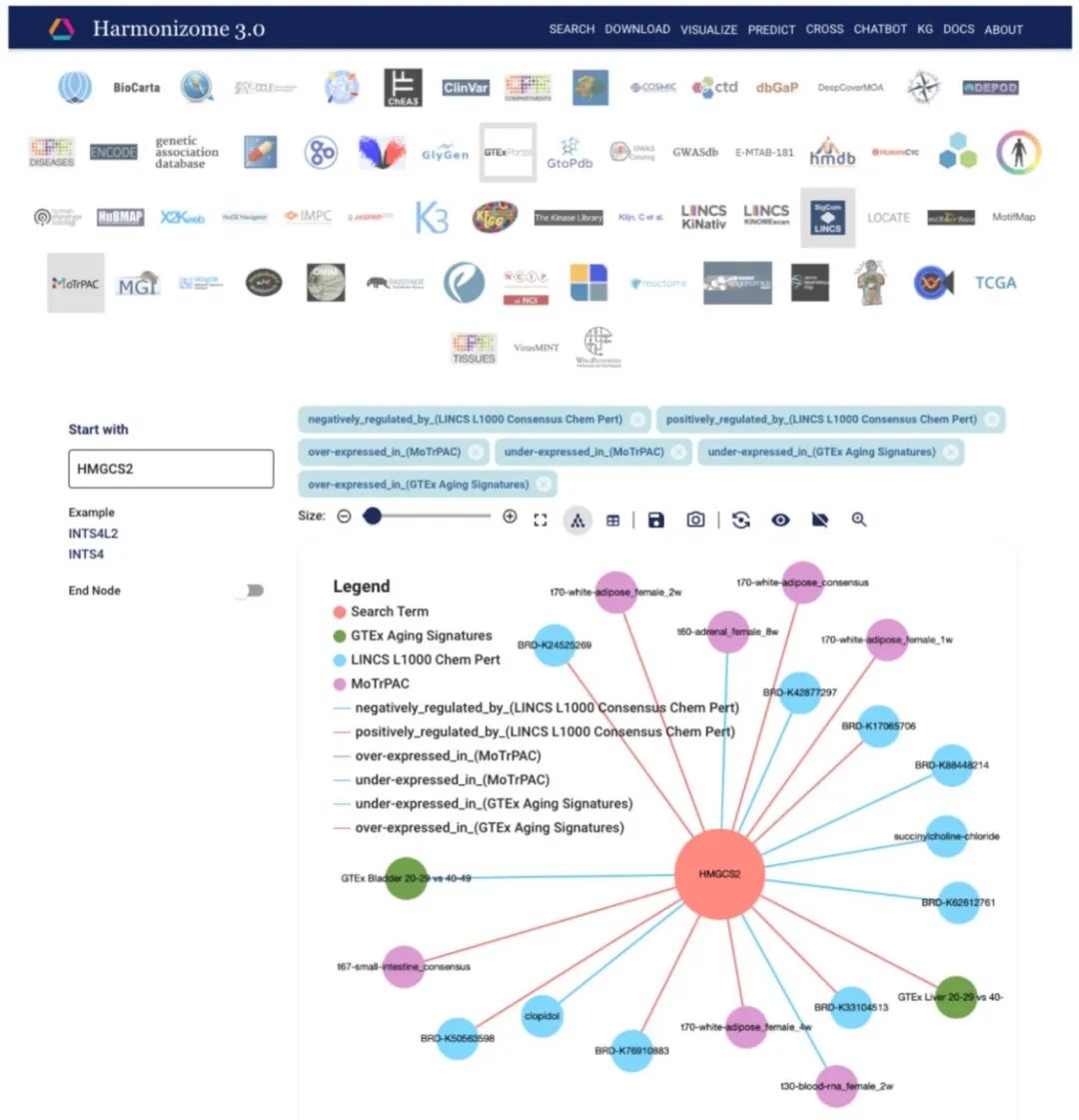
图5 Harmonizome 3.0知识图谱的截图。基因与属性之间的关联以球棍图的形式可视化。查询的基因(HMGCS2)位于子网络图的中心。与HMGCS2相关的属性来自每个选定的Harmonizome 3.0数据集:GTEx组织特异性衰老特征、LINCS L1000 CMap化学扰动共识特征和MoTrPAC大鼠耐力运动训练。边表示正或负效应。点击资源图标可选择其他数据集的关联。子网络可切换为使用力导向、层次或几何布局。可使用滑块调整显示的关联数量。还可选择将子网络以表格形式查看、保存为表格格式、导出子网络为图像、查看边标签以及图例。
参考
[1] Nucleic Acids Res. 2024 Nov 20:gkae1080. doi: 10.1093/nar/gkae1080
注:AI辅助翻译,如有错误欢迎指出。请以复制粘贴,附上本号名片的方式转载此文。


