全网目前最完整最详细的DTI提取结构矩阵教程,跟着一步步做就一定能做出来(注:没有任何套路,亲测过程ok,结果ok)_如何获取focus dti序列
前几天发布了一个基于fsl的生成纤维束及结构矩阵的文章,但是自己实际运行的结果总感觉不太好,所以又经过搜索整理发现,结合使用MRtrix3,ANTS和fsl三者效果会更加高效,那么也记录一下全部过程,想做的跟着下面一步步做一定可以做出来。(建议优先使用这一版,上一篇文章作为额外参考)
我这边先进行处理一个受试,所以文件就在一个文件夹下,省的路径麻烦,后面可能会有集成的脚本,但我还是觉得应该先顺着流程跟着处理走一遍,总归是有好处的。另外,还是做一步验证一步的影像,看看合不合适,合适再进行下一步,不合适的及时调整。
1.环境准备
老样子,我还是在windows的wsl2环境中运行的,需要下载fsl和MRtrix3还有ANTS,前两个可以自行搜索或者百度或者问gpt,应该都好安装,对于ANTS来说直接下载二进制文件进行编译会相对方便一些,链接如下:Releases · ANTsX/ANTs![]() https://github.com/ANTsX/ANTs/releases
https://github.com/ANTsX/ANTs/releases
安装完后可以自行测试一下是否安装成功,如果能显示版本号或者打开GUI界面那么就是成功了。
2.数据准备
老样子,需要准备DTI文件,T1文件,.bval和.bvec文件,如下图所示,为了方便,我就都放到一个文件夹里面了,运行下面的代码也方便。命名的话我都简化了,省的麻烦,dti和T1这种的。
3.DTI处理
终端cd至数据目录下,进行去噪,矫正Gibbs伪影和头动与涡流矫正:
dwidenoise dti.nii.gz dti_denoised.nii.gz ![]()
mrdegibbs dti_denoised.nii.gz dti_denoised_degibbs.nii.gz![]()
eddy_correct dti_denoised_degibbs.nii.gz dti_eddy.nii.gz 0![]()
接下来需要提取b0图像:
fslroi dti_eddy.nii.gz b0.nii.gz 0 1![]()
去除b0图像颅骨,提取脑组织:
bet b0.nii.gz b0_brain.nii.gz -m -f 0.2
4.T1处理
首先进行脑提取:
bet T1.nii.gz T1_brain.nii.gz -m -f 0.3
生成5TT图像(5种组织类型):
5ttgen fsl T1_brain.nii.gz 5tt.mif -premasked![]()
5.DTI-T1配准(使用ANTS)
刚性配准(fsl)
flirt -in b0_brain.nii.gz -ref T1_brain.nii.gz -omat diff2anat.mat -dof 6 -v![]()
非线性配准(ANTS)
antsRegistrationSyN.sh -d 3 -f T1_brain.nii.gz -m b0_brain.nii.gz -o diff2anat_ -t r
这步之后会生成一个.mat文件,也就diff2anat_0GenericAffine.mat这个文件,首先需要将这个.mat文件转换为.txt文本文件,代码如下:
ConvertTransformFile 3 diff2anat_0GenericAffine.mat diff2anat_0GenericAffine.txt然后打开.txt文件,我的是这样子的,如下所示(这一步有两种方法,这一步最简单的方法,下面还有精确的方法):
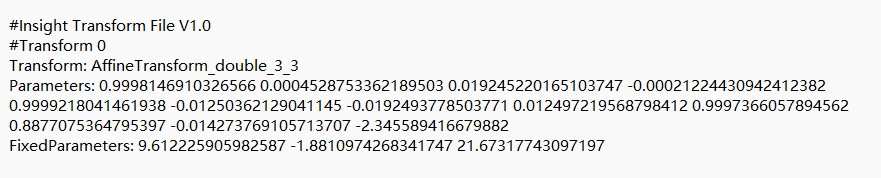
手动将第一行,第二行,第三行,最后一行都删掉,只保留Parameters这一行的数字,“Parameters:”也删掉。
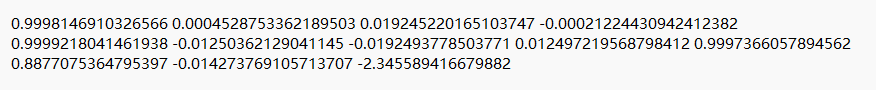
这里有12个数字或者16个数字都是正常的。(应该都是正常的,不正常自行gpt或者百度吧,我也不着知道咋办)
将三个数字分为一组,前三个数字放在第一排,再三个数字放在第二排,接下来三个数字放在第三排,如果是16个的话,那就再三个数字放在第四排,如果是12个那就都是0.0就好,将最后的三个或者4个数字放在每一行的最后一个,如果是三个数字的话最右下角的数字是1.0。
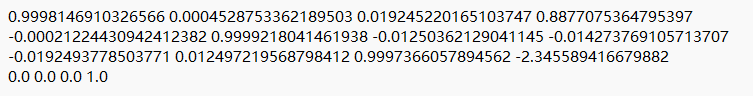
结果如上图,保存文件就ok了。
上面的方法是快速测试方法,如果条件允许,有python,可以运行以下脚本,可以获得更加具体且标准的最后一列的数值,后面的处理效果也会更好:
import numpy as np# 定义矩阵和向量R = np.array([ [0.9998146910326566, 0.0004528753362189503, 0.019245220165103747], [-0.00021224430942412382, 0.9999218041461938, -0.01250362129041145], [-0.0192493778503771, 0.012497219568798412, 0.9997366057894562]])T_initial = np.array([0.8877075364795397, -0.014273769105713707, -2.345589416679882])C = np.array([9.612225905982587, -1.8810974268341747, 21.67317743097197])# 计算 R × CR_times_C = R.dot(C)# 计算 T_final = T_initial + C - R × CT_final = T_initial + C - R_times_C # 结果保留6位小数T_final = np.round(T_final, 6)数值都是.txt中的数值,没看懂的话可以多对照txt看几遍,看多了就明白了。
结果如下,六位小数就足够了:

保存文件。
![]()
然后将5TT图像映射到DTI空间:
mrtransform 5tt.mif -linear diff2anat_0GenericAffine.txt -inverse -interp linear -template b0_brain.nii.gz 5tt_in_dti.mif![]()
6.纤维追踪
首先将dti_eddy文件的后缀由.nii.gz修改为.mif格式,需要提供bvec和bval:
mrconvert dti_eddy.nii.gz dti_eddy.mif -fslgrad dti.bvec dti.bval然后构建响应函数:
dwi2response dhollander dti_eddy.mif response_wm.txt response_gm.txt response_csf.txt -mask b0_brain_mask.nii.gz计算FOD:
dwi2fod msmt_csd dti_eddy.mif response_wm.txt wm_fod.mif -mask b0_brain_mask.nii.gz提取灰白质影像:
5tt2gmwmi 5tt_in_dti.mif gmwmi_seed.mif ![]()
纤维追踪,可以根据自己的需求和硬件水平自行修改参数:
tckgen wm_fod.mif tracks.tck -algorithm iFOD2 \\ -seed_gmwmi gmwmi_seed.mif -act 5tt_in_dti.mif \\ -backtrack -crop_at_gmwmi -maxlength 250 -select 1000000经过一段时间的等待,大功告成,以下就是纤维束文件。
可以进行可视化看看生成的效果怎么样:
mrview T1_brain.nii.gz -tractography.load tracks.tck接下来再生成连接矩阵之前,还需要将自定义的模板映射到个体空间,以便分割脑区,我用的是AAL116,那么就需要将AAL116模板适应个体受试影像,以便分割不同个体脑区,以下是具体处理步骤。
7.生成MNI到T1以及T1到DTI配准文件
线性配准(生成仿射矩阵):
flirt -in ${FSLDIR}/data/standard/MNI152_T1_1mm_brain.nii.gz \\ -ref T1_brain.nii.gz \\ -omat MNI_to_T1_affine.mat \\ -dof 12 -cost mutualinfo \\ -out MNI-TEST.nii.gz
非线性配准(生成形变场):
fnirt --in=${FSLDIR}/data/standard/MNI152_T1_1mm_brain.nii.gz \\ --ref=T1_brain.nii.gz \\ --aff=MNI_to_T1_affine.mat \\ --cout=MNI_to_T1_warp.nii.gz \\ --iout=MNI_warped.nii.gzT1到DTI的线性配准:
flirt -in T1_brain.nii.gz \\ -ref b0_brain.nii.gz \\ -omat T1_to_DTI.mat \\ -dof 6 -cost mutualinfo![]()
应用配准矩阵查看对齐效果:
applywarp -i T1_brain.nii.gz \\ -r b0_brain.nii.gz \\ --premat=T1_to_DTI.mat \\ -o T1_in_DTI.nii.gz可视化检查:
fsleyes b0_brain.nii.gz T1_in_DTI.nii.gz如果二者基本上对齐就可以了。
8.有关ALL模板的处理
将AAL模板从MNI空间配准到T1空间,需要手动准备自己需要配准的模板,我这里使用的是AAL116:
applywarp --ref=T1_brain.nii.gz \\ --in=AAL116_1mm.nii.gz \\ --premat=MNI_to_T1_affine.mat \\ --out=AAL_in_T1.nii.gz \\ --interp=nn ![]()
验证结果:
mrview T1_brain.nii.gz -overlay.load AAL_in_T1.nii.gz基本对齐在同一空间就可以。
将文件从.nii.gz转换为MRtrix格式:
mrconvert AAL_in_T1.nii.gz AAL_in_T1.mif![]()
并且需要将T1_to_DTI.mat文件转换为MRtrix3支持的格式:
transformconvert T1_to_DTI.mat T1_brain.nii.gz b0_brain.nii.gz flirt_import T1_to_DTI_mrtrix.mat![]()
将T1空间的AAL模板转换到DTI空间:
mrtransform AAL_in_T1.mif \\ -linear T1_to_DTI_mrtrix.mat \\ -template b0_brain.nii.gz \\ -interp nearest -datatype uint32 \\ AAL_in_DTI.mif验证结果:
mrview b0_brain.nii.gz -overlay.load AAL_in_DTI.mif如果完全贴合,就说明处理正确。
9.生成连接矩阵
以下代码生成的是归一化后的连接强度,有利于跨个体分析,组间比较等等。
tck2connectome tracks.tck AAL_in_DTI.mif connectivity.csv \\ -scale_invnodevol -stat_edge mean -zero_diagonal如果就想要原始连接数,那就是以下代码:
tck2connectome tracks.tck AAL_in_DTI.mif raw_connectivity.csv \\ -stat_edge sum -zero_diagonal以上生成的都不是对称矩阵,如果想生成对称矩阵,那么就在代码中再加入-symmetric这个代码,就会生成对称矩阵了。
总结:
以上就是我从头到尾一步一步处理后生成的文件,历时四天,但最终做出来还是挺有成就感的,创作不易,如果对大家有帮助的话希望大家可以点个赞支持一下,十分感谢!!!如果有哪里不懂的也可以互相交流共同进步!!!
目前美中不足的就是没有一键化的处理脚本,因为中间涉及自己修改文件数值之类的,我以后也会持续改进,争取做出一键化的处理脚本!!!
写在最后的话:一开始的话我是准备上某鱼或者某某书想找代做,但是无奈收费太高,预算有限,只能硬着头皮一步一步做,但是自己做出来的喜悦还是难以言表的。最后希望大家以后都能积极创新,勇攀高峰!!!