基于Python+逻辑回归+XGBoost的肝癌预测项目,保存预测模型,基于streamlit快速搭建Web界面,输入患者特征并获取预测结果
一、前期准备:环境配置与数据加载
我用夸克网盘给你分享了「肝癌预测数据集和相关代码模型」,链接:https://pan.quark.cn/s/5ccf66c17f2d
kaggle源地址:https://www.kaggle.com/datasets/miadul/predict-liver-cancer-from-and-clinical-features

1. 依赖库
pandas/numpy:数据读取、清洗与数值计算;matplotlib/seaborn:数据可视化;scikit-learn:经典机器学习模型(逻辑回归、随机森林等)与评估工具;xgboost:高效的梯度提升树模型(适合医疗数据分类);shap:模型可解释性分析(医疗场景需明确“哪些特征导致癌症预测”)。
没有安装上述依赖库可以安装一下,例如:
pip install shap2. 加载数据并初步探查
通过pandas读取CSV文件,查看数据基本信息(维度、缺失值、数据类型),避免后续因数据格式问题报错:
import pandas as pdimport numpy as np# 加载数据(需替换为你的数据集本地路径)df = pd.read_csv(\"predict_liver_cancer.csv\")# 1. 查看数据前5行,了解特征分布print(\"数据前5行:\")print(df.head())# 2. 查看数据维度(行数×列数)print(f\"\\n数据维度:{df.shape}\") # 预期输出 (5000, 14)# 3. 检查缺失值(该数据集为合成数据,通常无缺失,但需验证)print(\"\\n各列缺失值数量:\")print(df.isnull().sum())# 4. 查看数据类型与统计描述print(\"\\n数据类型:\")print(df.dtypes)print(\"\\n数值型特征统计描述:\")print(df.describe()) # 仅显示age、bmi等数值列的均值、标准差、分位数等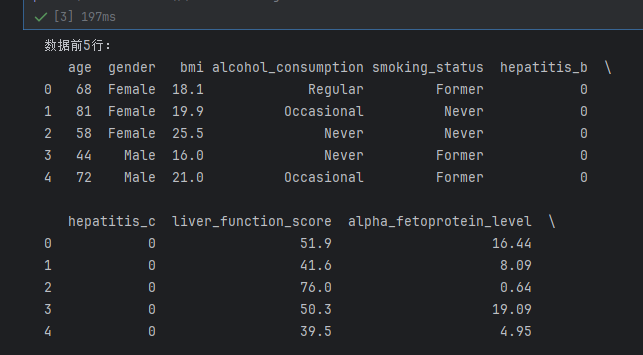
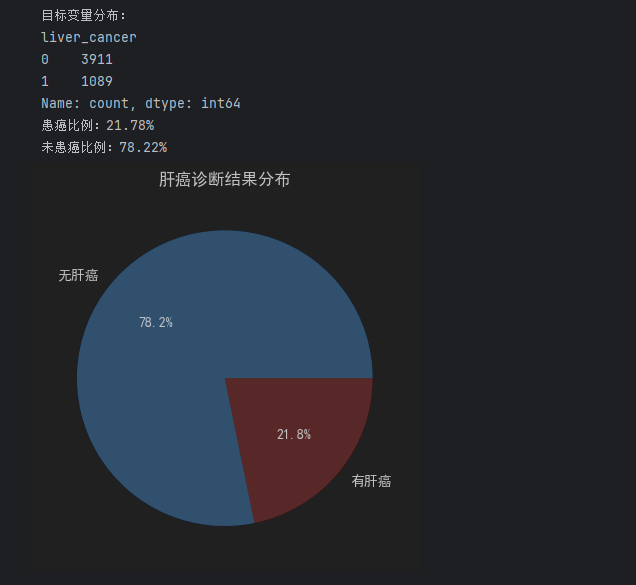
3. 目标变量分布检查
该任务为二分类问题(预测是否患肝癌:liver_cancer=1/0),需先确认正负样本是否平衡(避免模型偏向多数类):
# 统计目标变量数量与占比target_count = df[\"liver_cancer\"].value_counts()print(\"目标变量分布:\")print(target_count)print(f\"患癌比例:{target_count[1]/len(df):.2%}\")print(f\"未患癌比例:{target_count[0]/len(df):.2%}\")# 可视化目标变量分布(用饼图更直观)import matplotlib.pyplot as pltplt.pie(target_count, labels=[\"无肝癌\", \"有肝癌\"], autopct=\"%1.1f%%\", colors=[\"#66b3ff\", \"#ff9999\"])plt.title(\"肝癌诊断结果分布\")plt.show()- 如果两类样本比例差异过大(如患癌样本<10%),后续建模需用SMOTE过采样或类权重调整(如
class_weight=\'balanced\')解决 imbalance 问题。
二、核心环节1:探索性数据分析(EDA)
EDA是理解数据的关键,需从单特征分布、特征与目标的关联、特征间相关性三个维度展开,挖掘医疗数据中的潜在规律(如“肝炎患者是否更易患肝癌”“BMI与肝癌的关系”)。
1. 数值型特征分析(年龄、BMI、肝功能评分等)
重点查看数值特征的分布形态,以及不同肝癌诊断结果下的特征差异:
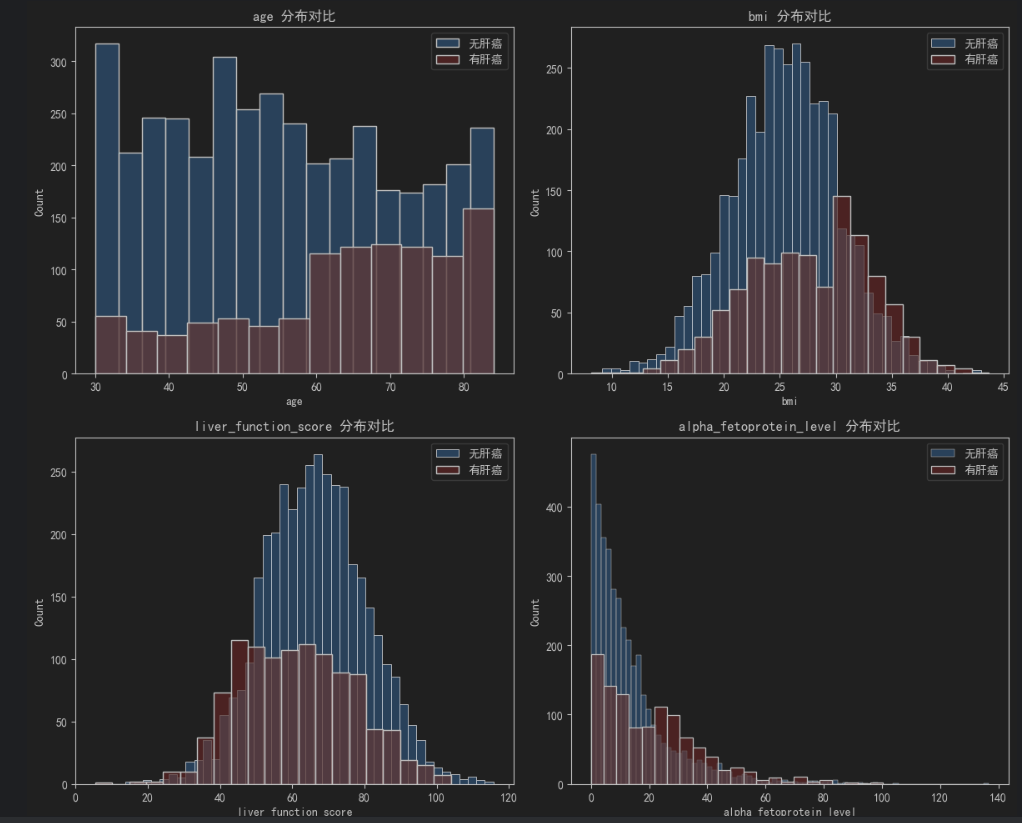
import seaborn as sns# 定义数值型特征列表(从数据集描述中提取)numeric_features = [\"age\", \"bmi\", \"liver_function_score\", \"alpha_fetoprotein_level\"]# 1. 绘制数值特征的直方图(按肝癌诊断分组,对比分布差异)fig, axes = plt.subplots(2, 2, figsize=(12, 10))axes = axes.flatten() # 展平子图数组,便于循环for i, feat in enumerate(numeric_features): # 无肝癌组 sns.histplot(df[df[\"liver_cancer\"]==0][feat], ax=axes[i], label=\"无肝癌\", color=\"#66b3ff\", alpha=0.7) # 有肝癌组 sns.histplot(df[df[\"liver_cancer\"]==1][feat], ax=axes[i], label=\"有肝癌\", color=\"#ff9999\", alpha=0.7) axes[i].set_title(f\"{feat} 分布对比\") axes[i].legend()plt.tight_layout()plt.show()# 2. 统计不同肝癌状态下数值特征的均值(医疗场景中“均值差异”有临床意义)numeric_stats = df.groupby(\"liver_cancer\")[numeric_features].mean()print(\"不同肝癌状态下数值特征的均值:\")print(numeric_stats)- 关键观察点:例如
alpha_fetoprotein_level(甲胎蛋白,临床肝癌标志物)在“有肝癌”组的均值应显著高于“无肝癌”组,若符合则数据逻辑合理。
2. 分类型特征分析(性别、饮酒、吸烟、肝炎史等)
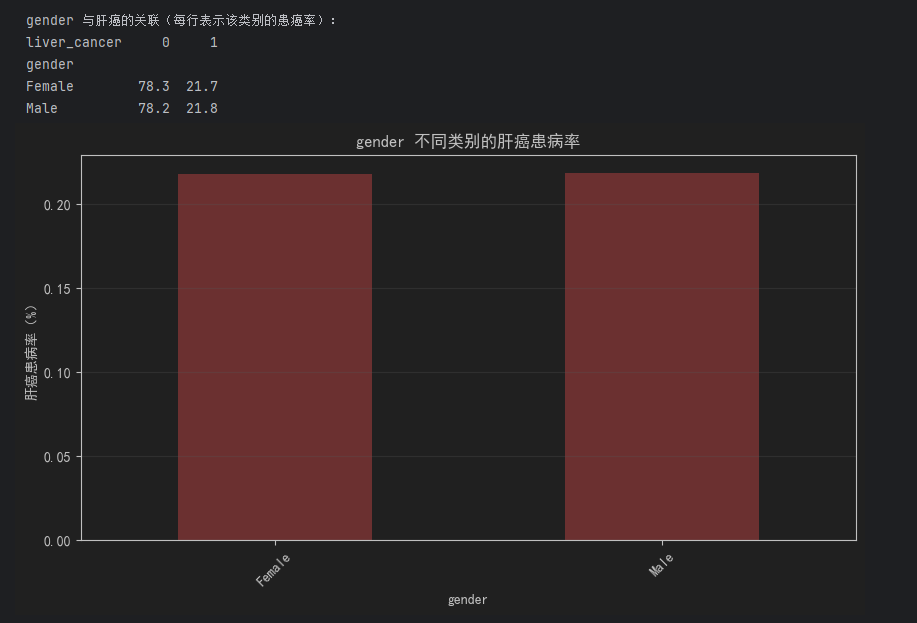
分类型特征需用交叉表或条形图分析其与肝癌的关联(如“男性vs女性的患癌率”“有乙肝vs无乙肝的患癌率”):
# 定义分类型特征列表(含二分类特征如hepatitis_b,及多分类如alcohol_consumption)cat_features = [\"gender\", \"alcohol_consumption\", \"smoking_status\", \"hepatitis_b\", \"hepatitis_c\", \"cirrhosis_history\", \"family_history_cancer\", \"physical_activity_level\", \"diabetes\"]# 循环分析每个分类型特征与肝癌的关联for feat in cat_features: # 1. 计算交叉表:特征类别 × 肝癌状态,得到数量分布 cross_tab = pd.crosstab(df[feat], df[\"liver_cancer\"], normalize=\"index\") # normalize=\"index\" 计算每行占比(即该类别的患癌率) print(f\"\\n{feat} 与肝癌的关联(每行表示该类别的患癌率):\") print(cross_tab.round(3) * 100) # 转为百分比,保留1位小数 # 2. 可视化:条形图展示不同类别的患癌率 plt.figure(figsize=(10, 5)) cross_tab[1].plot(kind=\"bar\", color=\"#ff6b6b\") # 只展示“患癌率”(liver_cancer=1的列) plt.title(f\"{feat} 不同类别的肝癌患病率\") plt.xlabel(feat) plt.ylabel(\"肝癌患病率(%)\") plt.xticks(rotation=45) plt.grid(axis=\"y\", alpha=0.3) plt.show()- 关键医疗逻辑验证:例如
cirrhosis_history(肝硬化史)、hepatitis_b(乙肝)组的患癌率应显著高于无病史组,若符合则数据符合医学常识。
3. 特征间相关性分析
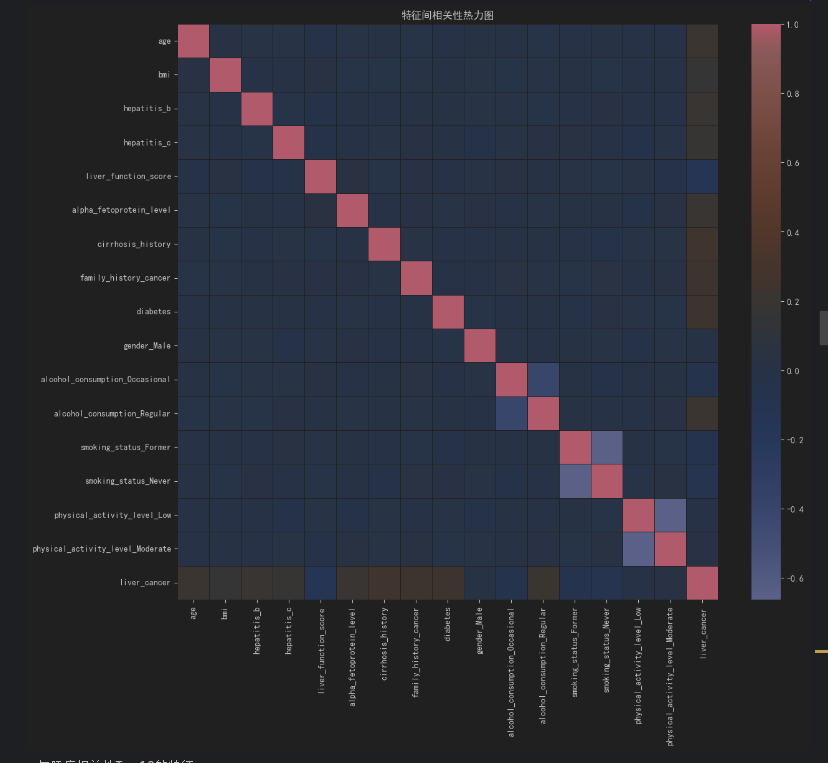
通过热力图查看特征间的线性相关程度,避免建模时的“多重共线性”问题(如两个高度相关的特征可能导致模型系数不稳定):
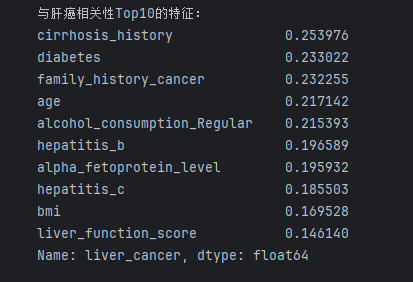
# 先将分类型特征转为数值型(one-hot编码),再计算相关系数df_encoded = pd.get_dummies(df.drop(\"liver_cancer\", axis=1), drop_first=True) # drop_first避免虚拟变量陷阱df_encoded[\"liver_cancer\"] = df[\"liver_cancer\"] # 加回目标变量# 计算Pearson相关系数corr_matrix = df_encoded.corr()# 可视化热力图(重点关注与目标变量的相关性)plt.figure(figsize=(14, 12))sns.heatmap(corr_matrix, annot=False, cmap=\"coolwarm\", fmt=\".2f\", linewidths=0.5)plt.title(\"特征间相关性热力图\")plt.show()# 提取与“肝癌”相关性Top10的特征(按绝对值排序)target_corr = corr_matrix[\"liver_cancer\"].drop(\"liver_cancer\").abs().sort_values(ascending=False)print(\"与肝癌相关性Top10的特征:\")print(target_corr.head(10))- 预期结果:
alpha_fetoprotein_level(甲胎蛋白)、cirrhosis_history(肝硬化史)、hepatitis_b/c(肝炎)应与目标变量相关性最高,符合临床认知。
三、核心环节2:数据预处理
医疗数据需处理分类型特征编码、数值型特征标准化、样本平衡,确保模型输入格式正确且训练稳定。
from sklearn.model_selection import train_test_splitfrom sklearn.preprocessing import StandardScalerfrom imblearn.over_sampling import SMOTE # 处理样本不平衡# 1. 分离特征(X)和目标变量(y)X = df.drop(\"liver_cancer\", axis=1)y = df[\"liver_cancer\"]# 2. 分类型特征编码(one-hot编码,适用于无顺序的类别如gender、alcohol_consumption)X_encoded = pd.get_dummies(X, drop_first=True) # drop_first=True:避免多重共线性(如Male=1则Female=0,无需保留Female列)# 3. 划分训练集和测试集(8:2分割,保证数据分布一致)X_train, X_test, y_train, y_test = train_test_split( X_encoded, y, test_size=0.2, random_state=42, stratify=y # stratify=y:按目标变量分布分层,保证训练/测试集患癌率一致)# 4. 数值型特征标准化(对逻辑回归、SVM等敏感模型必要,消除量纲影响)# 先筛选数值型特征列(从原始X中提取)numeric_cols = [\"age\", \"bmi\", \"liver_function_score\", \"alpha_fetoprotein_level\"]# 对应到编码后的X_train/X_test的列名(如无变化,直接用原列名)numeric_cols_encoded = [col for col in numeric_cols if col in X_train.columns]# 初始化标准化器,仅对数值列标准化scaler = StandardScaler()X_train[numeric_cols_encoded] = scaler.fit_transform(X_train[numeric_cols_encoded])X_test[numeric_cols_encoded] = scaler.transform(X_test[numeric_cols_encoded]) # 测试集用训练集的均值/标准差,避免数据泄露# 5. 处理样本不平衡(若之前发现患癌样本占比<15%,用SMOTE过采样)print(f\"过采样前训练集患癌样本占比:{y_train.sum()/len(y_train):.2%}\")smote = SMOTE(random_state=42)X_train_resampled, y_train_resampled = smote.fit_resample(X_train, y_train)print(f\"过采样后训练集患癌样本占比:{y_train_resampled.sum()/len(y_train_resampled):.2%}\") # 预期为50%四、核心环节3:机器学习建模与评估
医疗场景中,模型的召回率(Recall,避免漏诊)、精确率(Precision,避免误诊)、AUC(整体区分能力) 是关键指标。推荐从“简单基准模型”到“复杂模型”逐步迭代,对比效果。
1. 模型选择与训练
推荐4类适合医疗二分类的模型,覆盖不同复杂度:
以下是核心代码(以逻辑回归和XGBoost为例):
from sklearn.linear_model import LogisticRegressionfrom xgboost import XGBClassifierfrom sklearn.metrics import ( accuracy_score, precision_score, recall_score, f1_score, roc_auc_score, confusion_matrix, classification_report, roc_curve)# ---------------------- 1. 基准模型:逻辑回归 ----------------------lr_model = LogisticRegression(class_weight=\"balanced\", max_iter=1000, random_state=42)lr_model.fit(X_train_resampled, y_train_resampled)# 预测(输出概率,便于后续计算AUC)y_pred_lr = lr_model.predict(X_test)y_pred_proba_lr = lr_model.predict_proba(X_test)[:, 1] # 患癌的概率# ---------------------- 2. 高精度模型:XGBoost ----------------------xgb_model = XGBClassifier( objective=\"binary:logistic\", # 二分类任务 eval_metric=\"auc\", # 评估指标用AUC random_state=42, learning_rate=0.1, n_estimators=100)xgb_model.fit( X_train_resampled, y_train_resampled, eval_set=[(X_test, y_test)], # 验证集 early_stopping_rounds=10, # 早停:10轮无提升则停止,避免过拟合 verbose=False)# 预测y_pred_xgb = xgb_model.predict(X_test)y_pred_proba_xgb = xgb_model.predict_proba(X_test)[:, 1]2. 模型评估(医疗场景重点关注漏诊/误诊)
用混淆矩阵、分类报告、ROC曲线全面评估模型,尤其关注recall(漏诊率=1-recall,越低越好)和precision(误诊率=1-precision,越低越好):
# 定义评估函数,简化代码def evaluate_model(y_true, y_pred, y_pred_proba, model_name): print(f\"=== {model_name} 评估结果 ===\") # 混淆矩阵(TP:真阳性,TN:真阴性,FP:假阳性,FN:假阴性) cm = confusion_matrix(y_true, y_pred) print(\"混淆矩阵:\") print(cm) # 关键指标 print(f\"准确率(Accuracy):{accuracy_score(y_true, y_pred):.4f}\") print(f\"精确率(Precision):{precision_score(y_true, y_pred):.4f}\") # 预测为患癌的人中,实际患癌的比例(避免误诊) print(f\"召回率(Recall):{recall_score(y_true, y_pred):.4f}\") # 实际患癌的人中,被正确预测的比例(避免漏诊) print(f\"F1分数:{f1_score(y_true, y_pred):.4f}\") # 精确率和召回率的调和平均 print(f\"AUC值:{roc_auc_score(y_true, y_pred_proba):.4f}\") # 整体区分能力(0.9以上为优秀) # 分类报告(含每个类别的指标) print(\"分类报告:\") print(classification_report(y_true, y_pred)) # 绘制ROC曲线 fpr, tpr, _ = roc_curve(y_true, y_pred_proba) plt.plot(fpr, tpr, label=f\"{model_name} (AUC = {roc_auc_score(y_true, y_pred_proba):.4f})\", linewidth=2)# 评估两个模型evaluate_model(y_test, y_pred_lr, y_pred_proba_lr, \"逻辑回归\")evaluate_model(y_test, y_pred_xgb, y_pred_proba_xgb, \"XGBoost\")# 绘制ROC曲线对比plt.plot([0, 1], [0, 1], \"k--\", label=\"随机猜测(AUC=0.5)\")plt.xlabel(\"假阳性率(FPR)\")plt.ylabel(\"真阳性率(TPR)\")plt.title(\"肝癌预测模型ROC曲线对比\")plt.legend()plt.grid(alpha=0.3)plt.show()- 医疗场景优先级:若为“早期筛查”,需优先保证高召回率(避免漏诊潜在患者);若为“确诊辅助”,需平衡召回率和精确率(避免过度医疗)。
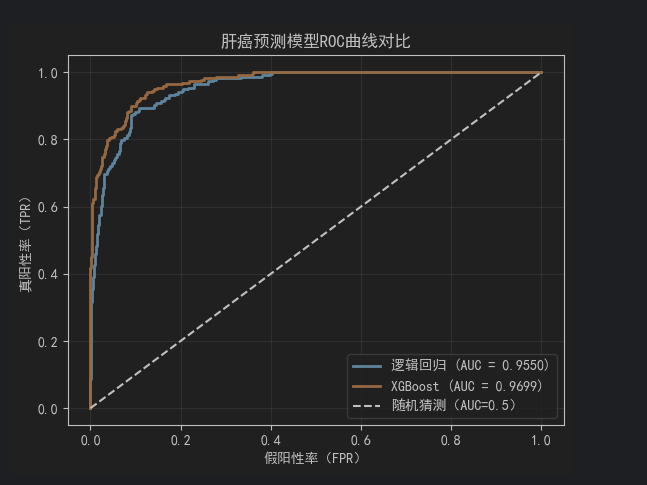
五、核心环节4:特征重要性与模型可解释性
医疗AI模型需“可解释”(医生需知道“为什么该患者被预测为患癌”),推荐用逻辑回归系数、随机森林/XGBoost特征重要性、SHAP值三种方式解读:
1. 逻辑回归系数(线性关系解读)
逻辑回归的系数正负表示“特征对患癌的影响方向”,绝对值表示影响强度:
# 提取逻辑回归系数,与特征名对应lr_coef = pd.DataFrame({ \"feature\": X_train.columns, \"coefficient\": lr_model.coef_[0] # 逻辑回归为二分类,系数数组长度=特征数})# 按系数绝对值排序,查看影响最大的特征lr_coef[\"abs_coef\"] = lr_coef[\"coefficient\"].abs()lr_coef_sorted = lr_coef.sort_values(\"abs_coef\", ascending=False)print(\"逻辑回归:影响肝癌的Top10特征(按系数绝对值):\")print(lr_coef_sorted.head(10)[[\"feature\", \"coefficient\"]])# 可视化Top10特征的系数(正负表示影响方向)plt.figure(figsize=(12, 6))sns.barplot(x=\"coefficient\", y=\"feature\", data=lr_coef_sorted.head(10))plt.title(\"逻辑回归:影响肝癌的Top10特征(系数)\")plt.xlabel(\"系数值(正=增加患癌风险,负=降低患癌风险)\")plt.ylabel(\"特征\")plt.grid(axis=\"x\", alpha=0.3)plt.show()- 预期结论:
cirrhosis_history_1(有肝硬化史)、hepatitis_b_1(有乙肝)的系数为正且绝对值大,说明这些特征会显著增加患癌风险。
2. XGBoost特征重要性(非线性关系解读)
XGBoost通过“特征在决策树中被使用的次数/增益”衡量重要性,适合解读非线性影响:
# 提取XGBoost特征重要性(两种指标:weight=使用次数,gain=信息增益)xgb_importance = pd.DataFrame({ \"feature\": X_train.columns, \"weight\": xgb_model.feature_importances_ # 默认是weight指标})# 按重要性排序xgb_importance_sorted = xgb_importance.sort_values(\"weight\", ascending=False)print(\"XGBoost:影响肝癌的Top10特征(按使用次数):\")print(xgb_importance_sorted.head(10))# 可视化Top10特征重要性plt.figure(figsize=(12, 6))sns.barplot(x=\"weight\", y=\"feature\", data=xgb_importance_sorted.head(10))plt.title(\"XGBoost:影响肝癌的Top10特征(重要性)\")plt.xlabel(\"特征重要性(weight指标)\")plt.ylabel(\"特征\")plt.grid(axis=\"x\", alpha=0.3)plt.show()3. SHAP值(个体样本解读)
SHAP值可量化“每个特征对单个患者预测结果的贡献”,医生可通过它解释“该患者患癌预测的依据”:
import shap# 初始化SHAP解释器(XGBoost专用)explainer = shap.TreeExplainer(xgb_model)# 计算测试集的SHAP值shap_values = explainer.shap_values(X_test)# 1. 总结图:全局特征影响(每个点代表一个样本的SHAP值,颜色代表特征值大小)plt.figure(figsize=(12, 8))shap.summary_plot(shap_values, X_test, feature_names=X_train.columns, plot_type=\"bar\")plt.title(\"SHAP值:全局特征重要性(Top15)\")plt.show()# 2. 个体解释图:以测试集中第1个样本为例,解读其预测依据sample_idx = 0 # 选择第1个测试样本plt.figure(figsize=(10, 6))shap.force_plot( explainer.expected_value, # 模型对所有样本的平均预测概率 shap_values[sample_idx], # 该样本的SHAP值 X_test.iloc[sample_idx], # 该样本的特征值 feature_names=X_train.columns, matplotlib=True)plt.title(f\"样本{sample_idx}的肝癌预测SHAP解释(红色=增加患癌风险,蓝色=降低风险)\")plt.show()- 解读逻辑:红色特征块表示“该特征值增加了该患者的患癌概率”(如
alpha_fetoprotein_level=80ng/mL,高于均值),蓝色块表示“降低风险”(如hepatitis_b=0,无乙肝)。
六、进阶操作:模型优化与部署
若需进一步提升模型性能或落地应用,可尝试以下方向:
1. 模型超参数优化
用GridSearchCV或RandomizedSearchCV调优参数(以XGBoost为例):
import pandas as pdimport numpy as npfrom sklearn.model_selection import RandomizedSearchCVfrom xgboost import XGBClassifierfrom imblearn.over_sampling import SMOTE# 假设X_train、X_test、y_train、y_test已完成前期预处理(编码、标准化)# 1. 处理数据类型和缺失值X_train = X_train.astype(\'float64\').fillna(0)X_test = X_test.astype(\'float64\').fillna(0)# 2. 重新过采样,确保X和y样本数匹配smote = SMOTE(random_state=42)X_train_resampled, y_train_resampled = smote.fit_resample(X_train, y_train)# 3. 定义XGBoost基础模型(不含搜索参数)xgb_model = XGBClassifier( objective=\"binary:logistic\", eval_metric=\"auc\", random_state=42)# 4. 定义参数搜索空间param_grid = { \"learning_rate\": [0.01, 0.1, 0.2], \"n_estimators\": [50, 100, 200], \"max_depth\": [3, 5, 7], \"subsample\": [0.8, 1.0], \"colsample_bytree\": [0.8, 1.0]}# 5. 随机搜索random_search = RandomizedSearchCV( estimator=xgb_model, param_distributions=param_grid, n_iter=20, cv=5, scoring=\"roc_auc\", random_state=42, verbose=1)# 6. 训练(此时应无报错)random_search.fit(X_train_resampled, y_train_resampled)# 后续操作...print(\"XGBoost最优参数:\", random_search.best_params_)best_xgb_model = random_search.best_estimator_y_pred_best_xgb = best_xgb_model.predict(X_test)2. 模型部署(简易Web应用)
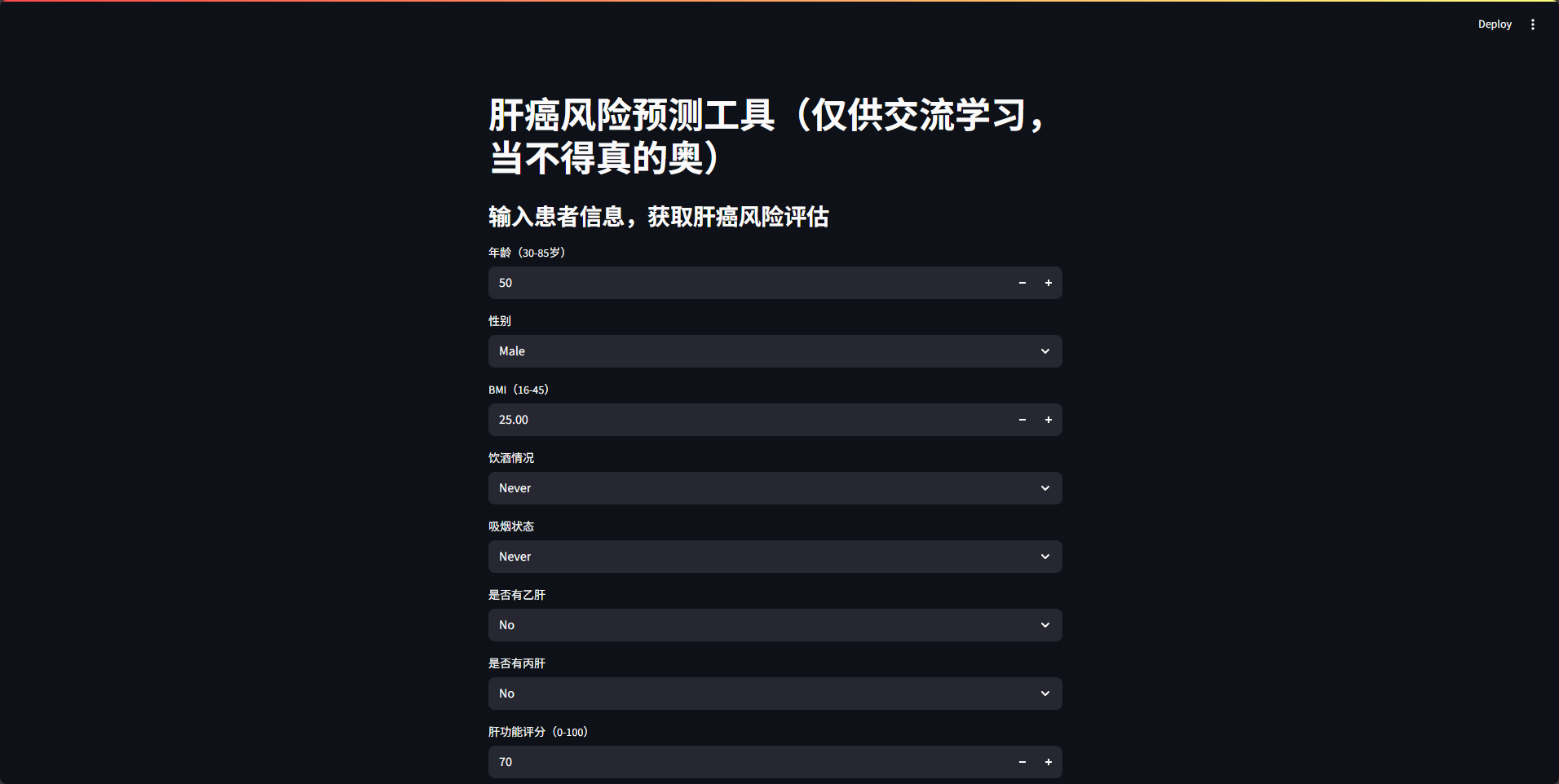
用Streamlit快速搭建Web界面,供医生输入患者特征并获取预测结果:
# 安装Streamlit:pip install streamlit# 保存为app.py,运行:streamlit run app.pyimport streamlit as stimport pandas as pd# 加载训练好的模型和标准化器(需提前用joblib保存)import joblibjoblib.dump(best_xgb_model, \"liver_cancer_xgb_model.pkl\")joblib.dump(scaler, \"scaler.pkl\")joblib.dump(X_train.columns, \"feature_columns.pkl\")# 加载模型model = joblib.load(\"liver_cancer_xgb_model.pkl\")scaler = joblib.load(\"scaler.pkl\")feature_cols = joblib.load(\"feature_columns.pkl\")# Web界面设计st.title(\"肝癌风险预测工具\")st.subheader(\"输入患者信息,获取肝癌风险评估\")# 1. 收集用户输入(对应数据集的13个特征)age = st.number_input(\"年龄(30-85岁)\", min_value=30, max_value=85, value=50)gender = st.selectbox(\"性别\", [\"Male\", \"Female\"])bmi = st.number_input(\"BMI(16-45)\", min_value=16.0, max_value=45.0, value=25.0)alcohol = st.selectbox(\"饮酒情况\", [\"Never\", \"Occasional\", \"Regular\"])smoking = st.selectbox(\"吸烟状态\", [\"Never\", \"Former\", \"Current\"])hepatitis_b = st.selectbox(\"是否有乙肝\", [\"No\", \"Yes\"])hepatitis_c = st.selectbox(\"是否有丙肝\", [\"No\", \"Yes\"])liver_score = st.number_input(\"肝功能评分(0-100)\", min_value=0, max_value=100, value=70)afp = st.number_input(\"甲胎蛋白水平(ng/mL)\", min_value=0.0, value=10.0)cirrhosis = st.selectbox(\"是否有肝硬化史\", [\"No\", \"Yes\"])family_cancer = st.selectbox(\"是否有癌症家族史\", [\"No\", \"Yes\"])activity = st.selectbox(\"体力活动水平\", [\"Low\", \"Moderate\", \"High\"])diabetes = st.selectbox(\"是否有糖尿病\", [\"No\", \"Yes\"])# 2. 将输入转为DataFrame,与训练数据格式一致input_data = pd.DataFrame({ \"age\": [age], \"gender\": [gender], \"bmi\": [bmi], \"alcohol_consumption\": [alcohol], \"smoking_status\": [smoking], \"hepatitis_b\": [1 if hepatitis_b==\"Yes\" else 0], \"hepatitis_c\": [1 if hepatitis_c==\"Yes\" else 0], \"liver_function_score\": [liver_score], \"alpha_fetoprotein_level\": [afp], \"cirrhosis_history\": [1 if cirrhosis==\"Yes\" else 0], \"family_history_cancer\": [1 if family_cancer==\"Yes\" else 0], \"physical_activity_level\": [activity], \"diabetes\": [1 if diabetes==\"Yes\" else 0]})# 3. 预处理输入数据(与训练流程一致)input_encoded = pd.get_dummies(input_data, drop_first=True)# 确保输入特征与训练集完全一致(补充缺失的虚拟变量列,值为0)for col in feature_cols: if col not in input_encoded.columns: input_encoded[col] = 0input_encoded = input_encoded[feature_cols] # 调整列顺序# 4. 标准化数值特征input_encoded[numeric_cols_encoded] = scaler.transform(input_encoded[numeric_cols_encoded])# 5. 预测并展示结果if st.button(\"预测肝癌风险\"): prob = model.predict_proba(input_encoded)[0, 1] # 患癌概率 st.subheader(f\"预测结果:\") if prob >= 0.5: st.warning(f\"该患者患肝癌的概率为 {prob:.2%},建议进一步临床检查!\") else: st.success(f\"该患者患肝癌的概率为 {prob:.2%},风险较低。\")七、总结与注意事项
- 数据特性适配:该数据集为合成数据,无真实患者隐私问题,可放心用于实验,但实际医疗场景需严格遵守HIPAA、隐私保护法等规范。
- 医疗逻辑优先:建模过程中需结合医学常识(如甲胎蛋白、肝炎史是肝癌关键风险因素),若特征重要性与临床认知冲突,需检查数据或模型问题。
- 模型局限性:机器学习模型仅为“辅助工具”,不能替代医生诊断,需在结果中明确标注“预测结果仅供参考,以临床检查为准”。
八、如何运行streamlit快速搭建Web界面
1、下载运行文件和模型文件
先把下面的这个文件下载下来,这是在标题一里呢。
2、进入你存放项目文件的文件夹
点击右侧控制台,cd到你存放文件的地方
3、输入以下命令运行
streamlit run app.py