从0开始学习R语言--Day60--EM插补法
虽然我们常常在数据处理中做数据分布以及异常值的处理,但实际上对于缺失值,很多时候我们都不能简单地去删除或赋予0值,毕竟很多都是有意义的数据,只是可能因为各种原因没有在数据面板中显示,直接删除或赋予0这种忽略数据本身意义的做法,会破坏数据的属性,扭曲数据的性质。
一般来说,对于各种缺失的数据,我们都会用EM插补法来填补数据,原理是根据缺失所属的数据列,粗略估计一个数据后,计算等于这个值的概率,然后重复这个过程指到数值不再发生变化,简单来说就是根据已有的数据列参考来回归一个数据。
以下是一个例子:
# 加载必要的包install.packages(\'mice\',type = \'binary\')library(mice) # 提供EM插补功能library(mvtnorm) # 用于生成多元正态分布数据library(norm) # 提供EM算法实现# 1. 生成模拟数据集set.seed(123)n <- 200 # 样本量p <- 5 # 变量数# 生成完整的多元正态分布数据mu <- c(5, 10, 15, 20, 25) # 均值向量sigma <- matrix(c( # 协方差矩阵 4, 2, 1, 0.5, 0.1, 2, 9, 3, 1, 0.5, 1, 3, 16, 4, 1, 0.5, 1, 4, 25, 5, 0.1, 0.5, 1, 5, 36), ncol = p)complete_data <- rmvnorm(n, mean = mu, sigma = sigma)colnames(complete_data) <- paste0(\"X\", 1:p)# 2. 人为制造缺失值 (MCAR机制)missing_data <- complete_datafor (j in 1:p) { # 每个变量随机缺失20% missing_indices <- sample(1:n, size = n * 0.2) missing_data[missing_indices, j] <- NA}# 查看缺失模式summary(missing_data)md.pattern(missing_data)# 3. 使用norm包进行EM插补# 首先需要对数据进行预处理s <- prelim.norm(missing_data) # 预处理thetahat <- em.norm(s) # EM算法估计参数# 获取插补后的数据集em_imputed <- imp.norm(s, thetahat, missing_data)# 查看插补后的完整数据集head(em_imputed)# 4. 使用mice包进行EM插补 (更简单的方法)em_mice <- mice(missing_data, method = \"norm\", m = 1, maxit = 50)complete_mice <- complete(em_mice)# 比较原始数据和插补数据par(mfrow = c(2, 3))for (i in 1:p) { plot(density(complete_data[, i], na.rm = TRUE), main = paste(\"X\", i), col = \"blue\") lines(density(complete_mice[, i], na.rm = TRUE), col = \"red\") legend(\"topright\", legend = c(\"Original\", \"Imputed\"), col = c(\"blue\", \"red\"), lty = 1)}# 5. 评估插补质量# 计算均方误差 (仅对缺失部分)mse <- numeric(p)for (i in 1:p) { missing_ind <- is.na(missing_data[, i]) mse[i] <- mean((complete_data[missing_ind, i] - complete_mice[missing_ind, i])^2)}print(paste(\"MSE for each variable:\", paste(round(mse, 3), collapse = \", \")))输出:
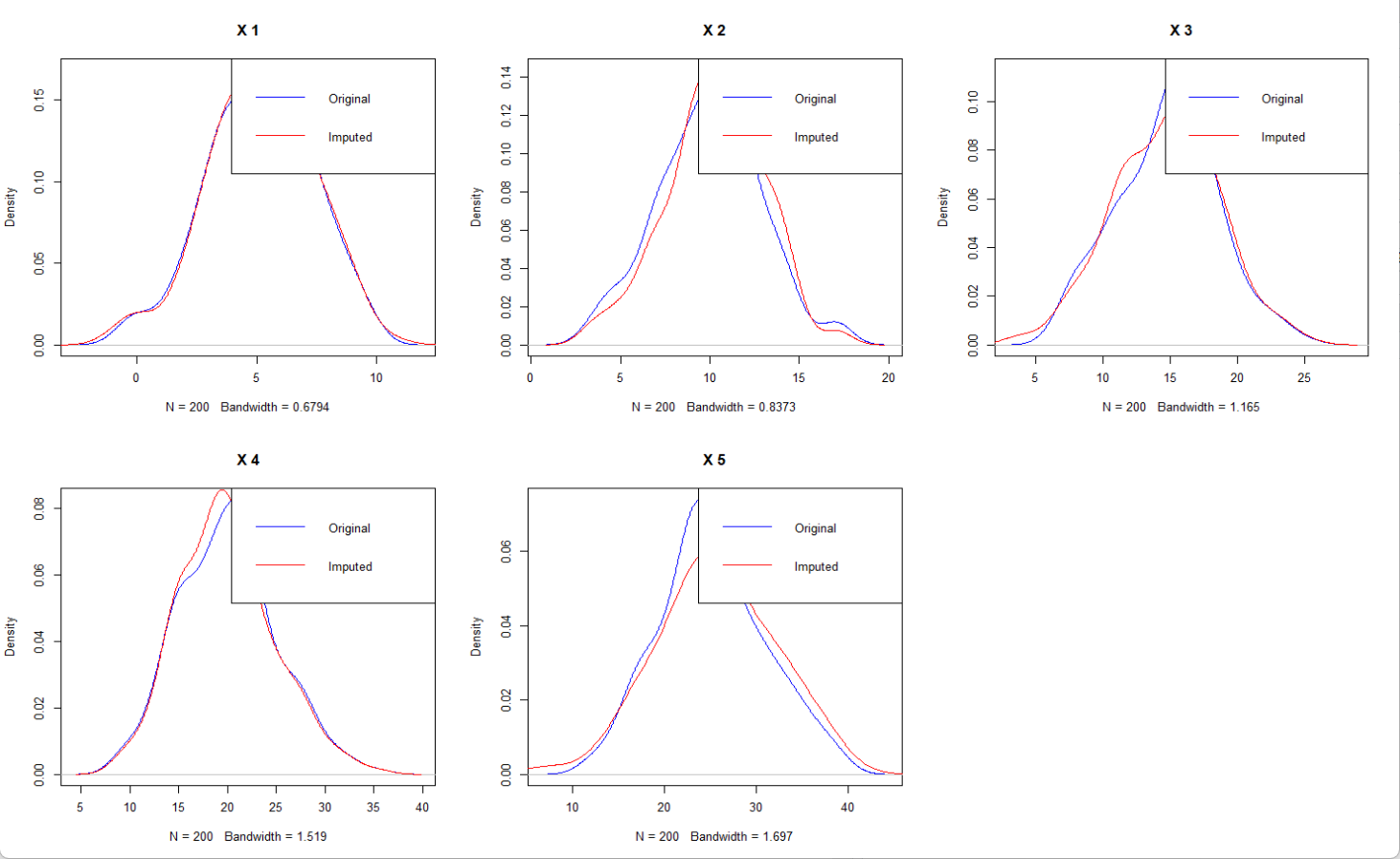
从整体来看,插补前后的曲线重合的地方很多,表明能够较好地修复数据,尤其是插补后没有看到有新的峰值或极端值。注意,如果缺失的值是一整年,同年份没有参考数据的话,就不能用EM,这种情况下的插补本质是一种预测了,要用函数的方法来做。


