AI大模型实践项目:医学影像分类器(肺结节检测)

🧑 博主简介:CSDN博客专家、CSDN平台优质创作者,高级开发工程师,数学专业,10年以上C/C++, C#,Java等多种编程语言开发经验,拥有高级工程师证书;擅长C/C++、C#等开发语言,熟悉Java常用开发技术,能熟练应用常用数据库SQL server,Oracle,mysql,postgresql等进行开发应用,熟悉DICOM医学影像及DICOM协议,业余时间自学JavaScript,Vue,qt,python等,具备多种混合语言开发能力。撰写博客分享知识,致力于帮助编程爱好者共同进步。欢迎关注、交流及合作,提供技术支持与解决方案。\\n技术合作请加本人wx(注明来自csdn):xt20160813
AI大模型实践项目:医学影像分类器(肺结节检测)
本项目利用深度学习技术开发肺结节检测分类器,基于 CT 影像区分良性和恶性结节,聚焦 卷积神经网络(CNN)、视觉变换器(Vision Transformer, ViT) 以及受 Med-PaLM 启发的多模态方法。使用 LUNA16 数据集,整合历史对话中的 Transformer 原理(自注意力、位置编码),增强代码支持 3D 处理和分割任务,新增高级可视化和隐私保护技术(如联邦学习)。文章结构如下:
- 项目概述:目标、数据集、技术栈和挑战。
- 理论基础:CNN、ViT、多模态模型及数学推导。
- 数据预处理:LUNA16 处理、3D CT 处理、数据增强。
- 模型实现:ResNet-50(3D)、ViT(LoRA)、多模态融合、分割任务。
- 评估与优化:交叉验证、评估指标、高级优化技术。
- 应用与展望:多模态融合、实时诊断、联邦学习。
一、项目概述
1.1 项目目标
- 功能:构建分类器,检测 CT 影像中的肺结节(良性/恶性)。
- 医学意义:肺结节是肺癌早期标志,自动分类可辅助诊断,降低漏诊率。
- 技术目标:
- 掌握深度学习工作流:数据预处理、模型训练、评估。
- 实现高召回率(Recall),减少假阴性(漏诊)。
- 比较 CNN、ViT 和多模态模型在医学影像中的性能。
- 提供可解释性(如 Grad-CAM),增强医生信任。
1.2 数据集
- LUNA16(Lung Nodule Analysis 2016):
- 包含 888 个 CT 扫描,标注肺结节位置和类别(良性/恶性)。
- 格式:DICOM(医学影像标准格式),3D 影像(512×512×N 片)。
- 下载:https://luna16.grand-challenge.org/
- RSNA(Radiological Society of North America):
- 提供胸部 CTA 影像,适合验证模型泛化性。
- 下载:https://www.rsna.org/
- 数据挑战:
- 类不平衡:恶性结节样本少(约 10-20%)。
- 高维数据:3D CT 需降维或分块处理。
- 噪声与伪影:CT 影像可能包含扫描噪声或金属伪影。
- 隐私保护:需符合《个人信息保护法》和 HIPAA/GDPR。
1.3 技术栈
- PyTorch:灵活实现 CNN、ViT 和 3D 模型。
- Hugging Face:提供预训练 ViT 和多模态模型支持。
- pydicom:读取和处理 DICOM 格式 CT 影像。
- MONAI:医学影像专用框架,支持 3D 数据处理和分割。
- scikit-learn/seaborn:评估指标(混淆矩阵、ROC 曲线)和可视化。
- Chart.js:性能对比图表。
- Flower:联邦学习框架,支持隐私保护训练。
1.4 医学影像分类挑战
- 数据稀缺:高质量标注数据有限,需迁移学习或数据增强。
- 高召回需求:漏诊(假阴性)成本高,需优化召回率。
- 3D 数据复杂性:CT 体视显微镜数据需高效处理。
- 可解释性:模型预测需与医学知识一致,需 Grad-CAM 或注意力可视化。
- 计算成本:3D 模型和 ViT 训练需高性能 GPU(如 NVIDIA A100)。
- 伦理与法规:确保公平性,保护患者隐私,符合医疗标准。
二、理论基础
2.1 卷积神经网络(CNN)
- 架构:
- 卷积层:提取局部特征(如结节边缘、纹理)。
- 池化层:降维,保留关键信息。
- 残差连接(ResNet):通过 y=x+F(x)y = x + F(x)y=x+F(x) 缓解梯度消失。
- 3D CNN:扩展卷积核为 3D(如 3×3×3),直接处理 CT 体视显微镜数据。
- 数学基础:
- 卷积操作(2D):
Y(i,j)=∑m∑nX(i+m,j+n)⋅K(m,n)+b Y(i,j) = \\sum_m \\sum_n X(i+m, j+n) \\cdot K(m,n) + bY(i,j)=m∑n∑X(i+m,j+n)⋅K(m,n)+b- XXX: 输入影像,KKK: 卷积核,bbb: 偏置。
- 3D 卷积:
Y(i,j,k)=∑m∑n∑pX(i+m,j+n,k+p)⋅K(m,n,p)+b Y(i,j,k) = \\sum_m \\sum_n \\sum_p X(i+m, j+n, k+p) \\cdot K(m,n,p) + bY(i,j,k)=m∑n∑p∑X(i+m,j+n,k+p)⋅K(m,n,p)+b- 处理体视显微镜数据,捕捉空间特征。
- 损失函数:
L=−∑iyilog(y^i)+λ∑∣∣W∣∣22 L = -\\sum_i y_i \\log(\\hat{y}_i) + \\lambda \\sum ||W||_2^2L=−i∑yilog(y^i)+λ∑∣∣W∣∣22- 交叉熵损失 + L2 正则化,防止过拟合。
- 卷积操作(2D):
- 适用性:高效提取局部特征,适合小区域结节检测;3D CNN 适配体视显微镜数据。
2.2 Vision Transformer (ViT)
- 架构(结合历史对话中的 Transformer):
- 图像分块:将 CT 影像分割为 Patch(如 16×16),展平为向量序列。
- 位置编码:添加正弦位置编码,保留 Patch 空间信息:
Epos(i,2k)=sin(i/100002k/d),Epos(i,2k+1)=cos(i/100002k/d) E_{\\text{pos}}(i, 2k) = \\sin(i / 10000^{2k/d}), \\quad E_{\\text{pos}}(i, 2k+1) = \\cos(i / 10000^{2k/d})Epos(i,2k)=sin(i/100002k/d),Epos(i,2k+1)=cos(i/100002k/d) - Transformer 编码器:多头自注意力(Multi-Head Attention)捕捉 Patch 间全局依赖。
- 分类头:CLS Token 或全局池化输出分类结果。
- 数学基础:
- Patch 嵌入:
z0=[xclass;xp1WE;xp2WE;… ;xpNWE]+Epos z_0 = [x_{\\text{class}}; x_p^1 W_E; x_p^2 W_E; \\dots; x_p^N W_E] + E_{\\text{pos}}z0=[xclass;xp1WE;xp2WE;…;xpNWE]+Epos- xpix_p^ixpi: 第 iii 个 Patch,WEW_EWE: 嵌入矩阵。
- 自注意力:
Attention(Q,K,V)=softmax(QKTdk)V \\text{Attention}(Q, K, V) = \\text{softmax}\\left(\\frac{QK^T}{\\sqrt{d_k}}\\right)VAttention(Q,K,V)=softmax(dkQKT)V- Q,K,V∈RN×dkQ, K, V \\in \\mathbb{R}^{N \\times d_k}Q,K,V∈RN×dk,NNN: Patch 数量,dkd_kdk: 嵌入维度。
- 多头注意力(历史对话):
MultiHead(Q,K,V)=Concat(head1,…,headh)WO \\text{MultiHead}(Q, K, V) = \\text{Concat}(\\text{head}_1, \\dots, \\text{head}_h)W_OMultiHead(Q,K,V)=Concat(head1,…,headh)WO- headi=Attention(QWiQ,KWiK,VWiV)\\text{head}_i = \\text{Attention}(QW_i^Q, KW_i^K, VW_i^V)headi=Attention(QWiQ,KWiK,VWiV)。
- Patch 嵌入:
- 适用性:全局建模能力强,适合复杂影像特征;需大规模预训练。
2.3 多模态模型(受 Med-PaLM 启发)
- 架构:
- 影像模块:ViT 处理 CT 影像。
- 文本模块:BERT 处理临床报告(如病史)。
- 融合模块:跨模态注意力整合影像和文本特征。
- 数学基础:
- 跨模态注意力(历史对话):
Attention(Qtext,Kimage,Vimage)=softmax(QtextKimageTdk)Vimage \\text{Attention}(Q_{\\text{text}}, K_{\\text{image}}, V_{\\text{image}}) = \\text{softmax}\\left(\\frac{Q_{\\text{text}}K_{\\text{image}}^T}{\\sqrt{d_k}}\\right)V_{\\text{image}}Attention(Qtext,Kimage,Vimage)=softmax(dkQtextKimageT)Vimage - 联合损失:
L=αLclass+βLalign L = \\alpha L_{\\text{class}} + \\beta L_{\\text{align}}L=αLclass+βLalign- LclassL_{\\text{class}}Lclass: 分类损失,LalignL_{\\text{align}}Lalign: 影像-文本对齐损失(如 CLIP 损失)。
- 跨模态注意力(历史对话):
- 适用性:结合临床信息,提升诊断精度,适合综合诊断。
2.4 迁移学习与 LoRA
- 预训练:
- CNN:ImageNet 预训练 ResNet-50,学习通用视觉特征。
- ViT:ImageNet 或 CheXpert 预训练 ViT,适配医学影像。
- LoRA(低秩适配):
- 仅更新低秩矩阵 ΔW=BA\\Delta W = BAΔW=BA,减少微调参数量:
W′=W+ΔW,ΔW=BA,B∈Rd×r,A∈Rr×k W\' = W + \\Delta W, \\quad \\Delta W = BA, \\quad B \\in \\mathbb{R}^{d \\times r}, A \\in \\mathbb{R}^{r \\times k}W′=W+ΔW,ΔW=BA,B∈Rd×r,A∈Rr×k - 适合 LUNA16 小数据集,降低计算成本。
- 仅更新低秩矩阵 ΔW=BA\\Delta W = BAΔW=BA,减少微调参数量:
- 优势:加速训练,适配小数据集,减少过拟合。
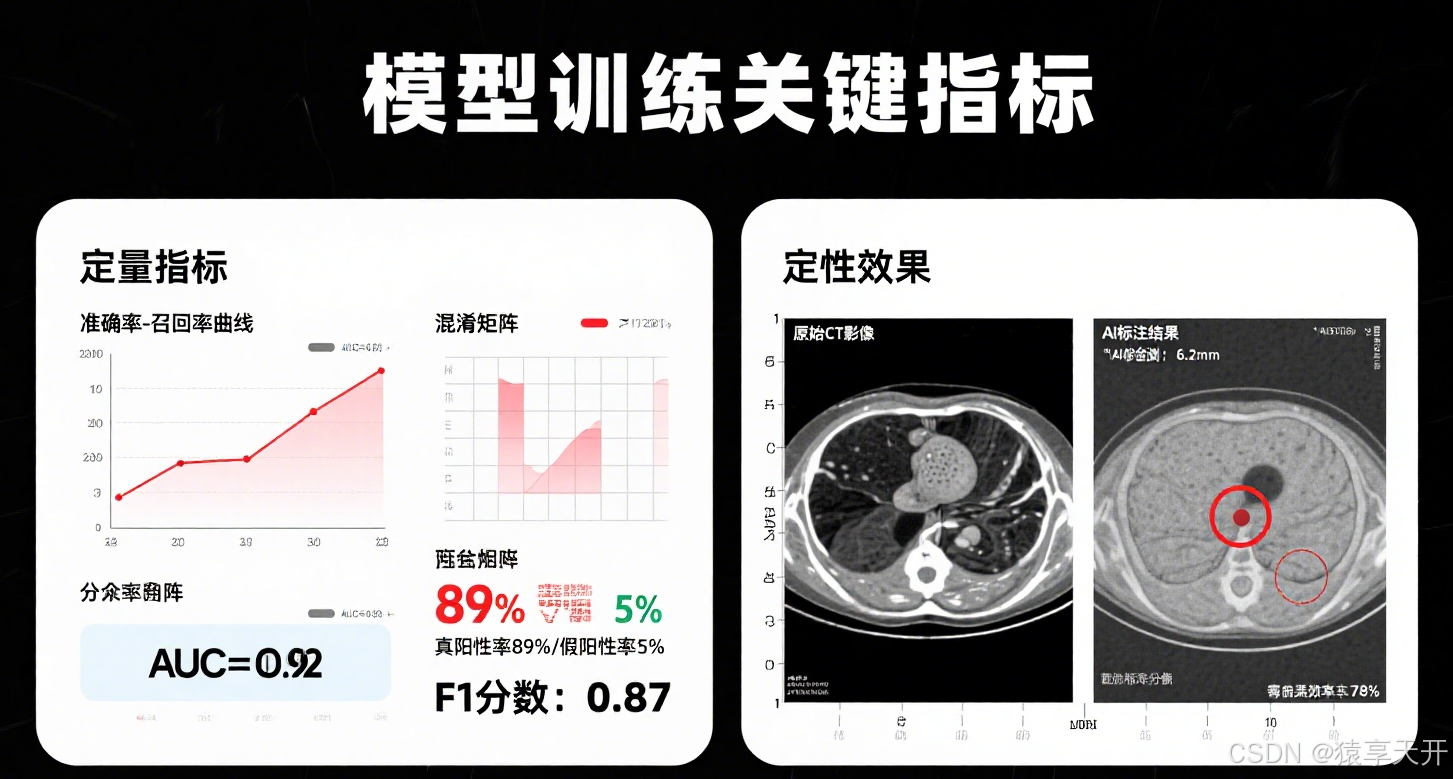
2.5 评估指标
- 混淆矩阵:计算真阳性(TP)、假阳性(FP)、真阴性(TN)、假阴性(FN)。
- 指标:
- 准确率:Accuracy=TP+TNTP+TN+FP+FN\\text{Accuracy} = \\frac{TP+TN}{TP+TN+FP+FN}Accuracy=TP+TN+FP+FNTP+TN
- 精确率:Precision=TPTP+FP\\text{Precision} = \\frac{TP}{TP+FP}Precision=TP+FPTP
- 召回率:Recall=TPTP+FN\\text{Recall} = \\frac{TP}{TP+FN}Recall=TP+FNTP(医学中关键)。
- F1 分数:F1=2⋅Precision⋅RecallPrecision+Recall\\text{F1} = 2 \\cdot \\frac{\\text{Precision} \\cdot \\text{Recall}}{\\text{Precision} + \\text{Recall}}F1=2⋅Precision+RecallPrecision⋅Recall
- ROC 曲线与 AUC:绘制真阳性率(TPR)对假阳性率(FPR),AUC 量化区分能力。
- 可解释性:Grad-CAM 和注意力热图,突出模型关注的结节区域。
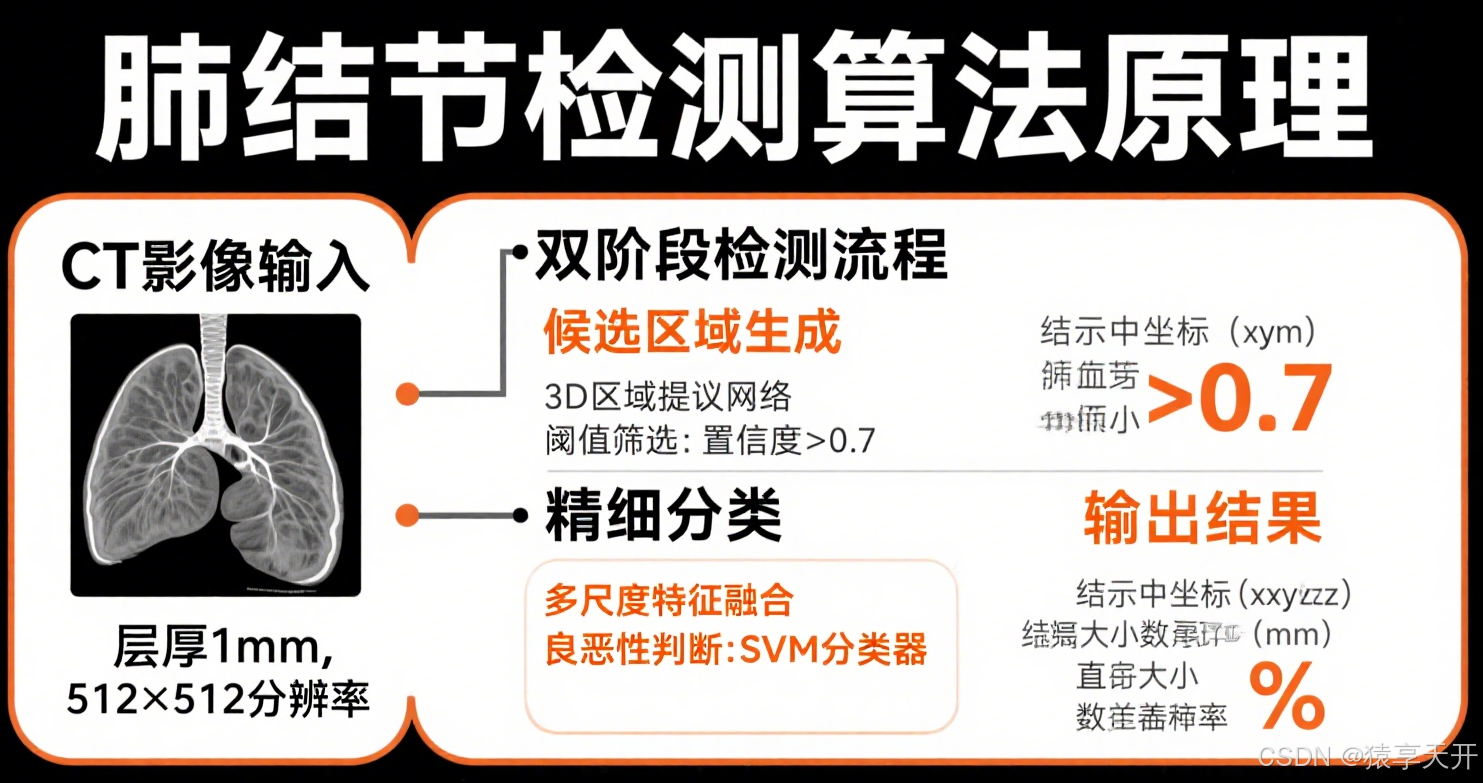
三、数据预处理
3.1 LUNA16 数据集处理
- 数据格式:DICOM 文件,3D CT 扫描(512×512×N 片)。
- 标注:CSV 文件,提供结节坐标(x, y, z)和类别(0: 良性,1: 恶性)。
- 预处理步骤:
- 读取 DICOM:使用 pydicom 加载 3D CT 影像。
- 归一化:将 Hounsfield 单位(HU)归一到 [0,1]:
Inorm=I−min(I)max(I)−min(I) I_{\\text{norm}} = \\frac{I - \\min(I)}{\\max(I) - \\min(I)}Inorm=max(I)−min(I)I−min(I) - 提取结节:基于坐标提取 3D 体视显微镜块(如 32×32×32)或 2D 切片。
- 数据增强:旋转、翻转、缩放、添加噪声,增加多样性。
- 数据集划分:80% 训练,10% 验证,10% 测试(分层确保类平衡)。
3.2 实现示例(Python)
以下为 LUNA16 数据预处理代码,支持 2D 和 3D 数据:
import pydicomimport numpy as npimport pandas as pdimport osfrom torch.utils.data import Datasetimport albumentations as Afrom albumentations.pytorch import ToTensorV2from monai.transforms import Compose, Resize, RandRotate, RandFlip, ToTensor# 自定义数据集class LUNA16Dataset(Dataset): def __init__(self, dicom_dir, annotations_file, mode=\'2d\', transform=None): \"\"\" LUNA16 数据集 :param dicom_dir: DICOM 文件目录 :param annotations_file: 标注 CSV 文件 :param mode: \'2d\' 或 \'3d\'(切片或体视显微镜) :param transform: 数据增强 \"\"\" self.dicom_dir = dicom_dir self.annotations = pd.read_csv(annotations_file) self.mode = mode self.transform = transform def __len__(self): return len(self.annotations) def __getitem__(self, idx): # 读取 DICOM dicom_id = self.annotations.iloc[idx][\'dicom_id\'] dicom_path = os.path.join(self.dicom_dir, dicom_id) ds = pydicom.dcmread(dicom_path) image = ds.pixel_array.astype(np.float32) # [H, W] 或 [D, H, W] # 归一化 image = (image - np.min(image)) / (np.max(image) - np.min(image) + 1e-6) # 提取结节区域 if self.mode == \'2d\': x, y, w, h, z = self.annotations.iloc[idx][[\'x\', \'y\', \'width\', \'height\', \'z\']].values image = image[z, y:y+h, x:x+w] # 2D 切片 else: # 3d x, y, z, w, h, d = self.annotations.iloc[idx][[\'x\', \'y\', \'z\', \'width\', \'height\', \'depth\']].values image = image[z:z+d, y:y+h, x:x+w] # 3D 体视显微镜块 # 数据增强 if self.transform: if self.mode == \'2d\': augmented = self.transform(image=image) image = augmented[\'image\'] else: image = self.transform(image[np.newaxis, ...])[0] # 添加通道维度 label = self.annotations.iloc[idx][\'label\'] # 0: 良性,1: 恶性 return {\'image\': image, \'label\': torch.tensor(label, dtype=torch.long)}# 数据增强transform_2d = A.Compose([ A.Resize(224, 224), A.Rotate(limit=30, p=0.5), A.HorizontalFlip(p=0.5), A.RandomBrightnessContrast(p=0.3), A.Normalize(mean=[0.5], std=[0.5]), ToTensorV2()])transform_3d = Compose([ Resize(spatial_size=(32, 32, 32)), RandRotate(range_x=30, prob=0.5), RandFlip(spatial_axis=0, prob=0.5), ToTensor()])# 加载数据集dataset_2d = LUNA16Dataset(dicom_dir=\'path/to/luna16\', annotations_file=\'annotations.csv\', mode=\'2d\', transform=transform_2d)dataset_3d = LUNA16Dataset(dicom_dir=\'path/to/luna16\', annotations_file=\'annotations.csv\', mode=\'3d\', transform=transform_3d)代码注释:
- pydicom:读取 DICOM 文件,提取像素数组。
- 模式选择:支持 2D 切片(224×224)和 3D 体视显微镜块(32×32×32)。
- 数据增强:
- 2D:旋转、翻转、亮度/对比度调整(albumentations)。
- 3D:体视显微镜旋转、翻转(MONAI)。
- 归一化:将 Hounsfield 单位归一到 [0,1]。
- 注意:需替换
dicom_dir和annotations_file为实际路径。
四、模型实现
4.1 CNN 实现(ResNet-50,3D 支持)
基于 ResNet-50,支持 2D 和 3D CT 影像分类:
import torchimport torch.nn as nnfrom torchvision.models import resnet50from monai.networks.nets import ResNetfrom torch.utils.data import DataLoaderfrom sklearn.metrics import accuracy_score, confusion_matrix# 3D ResNet-50class ResNet3D(nn.Module): def __init__(self, num_classes=2): super().__init__() self.resnet = ResNet(block=\'bottleneck\', layers=[3, 4, 6, 3], spatial_dims=3, n_input_channels=1, num_classes=num_classes) def forward(self, x): return self.resnet(x)# 2D ResNet-50class ResNet2D(nn.Module): def __init__(self, num_classes=2): super().__init__() self.resnet = resnet50(pretrained=True) self.resnet.conv1 = nn.Conv2d(1, 64, kernel_size=7, stride=2, padding=3) self.resnet.fc = nn.Linear(self.resnet.fc.in_features, num_classes) def forward(self, x): return self.resnet(x)# 训练函数def train_model(model, dataloader, criterion, optimizer, num_epochs=10, device=\'cuda\'): model = model.to(device) train_losses = [] for epoch in range(num_epochs): model.train() running_loss = 0.0 for batch in dataloader: images = batch[\'image\'].to(device) labels = batch[\'label\'].to(device) optimizer.zero_grad() outputs = model(images) loss = criterion(outputs, labels) loss.backward() optimizer.step() running_loss += loss.item() avg_loss = running_loss / len(dataloader) train_losses.append(avg_loss) print(f\'Epoch [{epoch+1}/{num_epochs}], Loss: {avg_loss:.4f}\') return train_losses# 数据加载器dataloader_2d = DataLoader(dataset_2d, batch_size=16, shuffle=True)dataloader_3d = DataLoader(dataset_3d, batch_size=8, shuffle=True)# 初始化模型device = torch.device(\'cuda\' if torch.cuda.is_available() else \'cpu\')model_2d = ResNet2D(num_classes=2)model_3d = ResNet3D(num_classes=2)criterion = nn.CrossEntropyLoss(weight=torch.tensor([0.3, 0.7]).to(device)) # 处理类不平衡optimizer_2d = torch.optim.Adam(model_2d.parameters(), lr=1e-4, weight_decay=1e-5)optimizer_3d = torch.optim.Adam(model_3d.parameters(), lr=1e-4, weight_decay=1e-5)# 训练train_losses_2d = train_model(model_2d, dataloader_2d, criterion, optimizer_2d, device=device)train_losses_3d = train_model(model_3d, dataloader_3d, criterion, optimizer_3d, device=device)# 推理def evaluate_model(model, dataloader, device=\'cuda\'): model.eval() predictions, true_labels = [], [] with torch.no_grad(): for batch in dataloader: images = batch[\'image\'].to(device) labels = batch[\'label\'].to(device) outputs = model(images) preds = torch.argmax(outputs, dim=1) predictions.extend(preds.cpu().numpy()) true_labels.extend(labels.cpu().numpy()) return predictions, true_labelspredictions_2d, true_labels_2d = evaluate_model(model_2d, dataloader_2d)predictions_3d, true_labels_3d = evaluate_model(model_3d, dataloader_3d)print(\"2D ResNet 准确率:\", accuracy_score(true_labels_2d, predictions_2d))print(\"3D ResNet 准确率:\", accuracy_score(true_labels_3d, predictions_3d))代码注释:
- 模型:2D ResNet-50(ImageNet 预训练)和 3D ResNet(MONAI 实现)。
- 损失函数:加权交叉熵,权重 [0.3, 0.7] 应对恶性结节稀缺。
- 优化器:Adam,学习率 1e-4,L2 正则化防止过拟合。
- 注意:3D 模型需更大显存(如 16GB),批大小减小至 8。
4.2 ViT 实现(Hugging Face,LoRA)
基于 ViT,结合 LoRA 微调,支持注意力可视化:
from transformers import ViTImageProcessor, ViTForImageClassificationfrom peft import LoraConfig, get_peft_modelfrom torch.utils.data import DataLoaderimport torchimport matplotlib.pyplot as plt# 加载 ViTprocessor = ViTImageProcessor.from_pretrained(\'google/vit-base-patch16-224\')model = ViTForImageClassification.from_pretrained(\'google/vit-base-patch16-224\', num_labels=2)# LoRA 微调lora_config = LoraConfig(r=8, lora_alpha=16, target_modules=[\"query\", \"value\"])model = get_peft_model(model, lora_config)# 训练设置device = torch.device(\'cuda\' if torch.cuda.is_available() else \'cpu\')model = model.to(device)criterion = nn.CrossEntropyLoss(weight=torch.tensor([0.3, 0.7]).to(device))optimizer = torch.optim.Adam(model.parameters(), lr=2e-5)# 训练dataloader = DataLoader(dataset_2d, batch_size=16, shuffle=True)train_losses = []for epoch in range(10): model.train() running_loss = 0.0 for batch in dataloader: images = batch[\'image\'].to(device) labels = batch[\'label\'].to(device) inputs = processor(images, return_tensors=\'pt\', do_rescale=False).to(device) outputs = model(**inputs).logits loss = criterion(outputs, labels) optimizer.zero_grad() loss.backward() optimizer.step() running_loss += loss.item() avg_loss = running_loss / len(dataloader) train_losses.append(avg_loss) print(f\'Epoch [{epoch+1}/10], Loss: {avg_loss:.4f}\')# 注意力可视化def visualize_attention(model, image, processor, device=\'cuda\'): model.eval() inputs = processor(image, return_tensors=\'pt\', do_rescale=False).to(device) with torch.no_grad(): outputs = model(**inputs, output_attentions=True) attentions = outputs.attentions[-1].mean(dim=1).squeeze(0) # 最后一层注意力 # 将注意力映射到原始图像 h, w = image.shape[-2:] attn_map = attentions.mean(dim=0).reshape(14, 14).cpu().numpy() # 假设 224/16=14 attn_map = np.resize(attn_map, (h, w)) plt.imshow(image.squeeze(0), cmap=\'gray\') plt.imshow(attn_map, cmap=\'jet\', alpha=0.5) plt.title(\'ViT 注意力热图\') plt.show()# 推理与评估predictions, true_labels = [], []with torch.no_grad(): for batch in dataloader: images = batch[\'image\'].to(device) labels = batch[\'label\'].to(device) inputs = processor(images, return_tensors=\'pt\', do_rescale=False).to(device) outputs = model(**inputs).logits preds = torch.argmax(outputs, dim=1) predictions.extend(preds.cpu().numpy()) true_labels.extend(labels.cpu().numpy())print(\"ViT 准确率:\", accuracy_score(true_labels, predictions))# 可视化示例sample_image = dataset_2d[0][\'image\']visualize_attention(model, sample_image, processor)代码注释:
- ViT:预训练 ViT-base,修改分类头为 2 类。
- LoRA:微调 query 和 value 矩阵,减少参数量。
- 注意力可视化:展示最后一层注意力热图,突出模型关注区域。
- 注意:仅支持 2D 影像,3D ViT 需扩展(见 4.4)。
4.3 多模态实现(受 Med-PaLM 启发)
结合 CT 影像和临床文本(如病史),实现多模态分类:
from transformers import ViTModel, BertTokenizer, BertModelimport torch.nn as nn# 多模态模型class MultiModalLungNoduleClassifier(nn.Module): def __init__(self, num_labels=2): super().__init__() self.vit = ViTModel.from_pretrained(\'google/vit-base-patch16-224\') self.bert = BertModel.from_pretrained(\'bert-base-uncased\') self.fusion = nn.Linear(768 + 768, 512) self.classifier = nn.Linear(512, num_labels) self.relu = nn.ReLU() self.dropout = nn.Dropout(0.1) def forward(self, image_inputs, text_inputs): vit_outputs = self.vit(**image_inputs).pooler_output # [batch, 768] bert_outputs = self.bert(**text_inputs).pooler_output # [batch, 768] combined = torch.cat((vit_outputs, bert_outputs), dim=-1) combined = self.relu(self.fusion(combined)) combined = self.dropout(combined) logits = self.classifier(combined) return logits# 数据集(扩展支持文本)class LUNA16MultiModalDataset(Dataset): def __init__(self, dicom_dir, annotations_file, texts, transform=None): self.dataset = LUNA16Dataset(dicom_dir, annotations_file, mode=\'2d\', transform=transform) self.texts = texts self.tokenizer = BertTokenizer.from_pretrained(\'bert-base-uncased\') def __getitem__(self, idx): item = self.dataset[idx] text = self.texts[idx] text_inputs = self.tokenizer(text, max_length=128, padding=\'max_length\', truncation=True, return_tensors=\'pt\') item[\'text_inputs\'] = {k: v.squeeze(0) for k, v in text_inputs.items()} return item def __len__(self): return len(self.dataset)# 数据准备(模拟临床文本)texts = [\"Patient with cough and fever, suspected malignancy.\"] * len(dataset_2d)multimodal_dataset = LUNA16MultiModalDataset(\'path/to/luna16\', \'annotations.csv\', texts, transform=transform_2d)dataloader = DataLoader(multimodal_dataset, batch_size=16, shuffle=True)# 训练model = MultiModalLungNoduleClassifier(num_labels=2).to(device)criterion = nn.CrossEntropyLoss(weight=torch.tensor([0.3, 0.7]).to(device))optimizer = torch.optim.Adam(model.parameters(), lr=2e-5)for epoch in range(10): model.train() running_loss = 0.0 for batch in dataloader: images = batch[\'image\'].to(device) labels = batch[\'label\'].to(device) image_inputs = processor(images, return_tensors=\'pt\', do_rescale=False).to(device) text_inputs = {k: v.to(device) for k, v in batch[\'text_inputs\'].items()} outputs = model(image_inputs, text_inputs) loss = criterion(outputs, labels) optimizer.zero_grad() loss.backward() optimizer.step() running_loss += loss.item() print(f\'Epoch [{epoch+1}/10], Loss: {running_loss/len(dataloader):.4f}\')代码注释:
- 模型:ViT(影像)+ BERT(文本),通过线性层融合特征。
- 数据:扩展 LUNA16 数据集,添加模拟临床文本。
- 注意:需真实临床文本(如病历),可从 MIMIC-III 获取。
4.4 分割任务(3D U-Net+ViT)
为肺结节分割,基于 MONAI 的 UNETR(U-Net+ViT):
from monai.networks.nets import UNETRfrom monai.data import DataLoader, Dataset as MonaiDatasetfrom monai.transforms import LoadImageD, EnsureChannelFirstD, Compose# 分割数据集transform_seg = Compose([ LoadImageD(keys=[\'image\']), EnsureChannelFirstD(keys=[\'image\']), Resize(spatial_size=(32, 32, 32)), ToTensor()])# 假设分割标注(mask)seg_data = [{\'image\': f\'path/to/luna16/{i}.dcm\', \'mask\': f\'path/to/mask/{i}.nii\'} for i in range(100)]seg_dataset = MonaiDataset(seg_data, transform=transform_seg)seg_dataloader = DataLoader(seg_dataset, batch_size=4, shuffle=True)# UNETR 模型model = UNETR(in_channels=1, out_channels=2, img_size=(32, 32, 32), feature_size=16).to(device)criterion = nn.CrossEntropyLoss()optimizer = torch.optim.Adam(model.parameters(), lr=1e-4)# 训练for epoch in range(10): model.train() running_loss = 0.0 for batch in seg_dataloader: images = batch[\'image\'].to(device) masks = batch[\'mask\'].to(device) outputs = model(images) loss = criterion(outputs, masks) optimizer.zero_grad() loss.backward() optimizer.step() running_loss += loss.item() print(f\'Epoch [{epoch+1}/10], Loss: {running_loss/len(seg_dataloader):.4f}\')代码注释:
- UNETR:结合 ViT 和 U-Net,处理 3D CT 分割。
- 数据:假设掩膜(mask)标注,需从 LUNA16 或 BraTS 获取。
- 注意:分割任务需更大显存(推荐 24GB)。
五、评估与优化
5.1 评估方法
- 交叉验证:5 折分层 K 折,确保类不平衡数据评估稳定。
- 混淆矩阵:计算 TP、FP、FN、TN,重点优化召回率。
- ROC 曲线与 AUC:评估模型区分能力。
- Dice 分数(分割任务):评估分割精度:
Dice=2∣P∩G∣∣P∣+∣G∣ \\text{Dice} = \\frac{2 |P \\cap G|}{|P| + |G|}Dice=∣P∣+∣G∣2∣P∩G∣- PPP: 预测掩膜,GGG: 真实掩膜。
5.2 实现示例(Python)
以下为分类和分割任务的评估代码:
from sklearn.metrics import confusion_matrix, roc_curve, auc, classification_reportfrom monai.metrics import DiceMetricimport seaborn as snsimport matplotlib.pyplot as plt# 分类评估def evaluate_classification(model, dataloader, processor=None, device=\'cuda\'): model.eval() predictions, true_labels, probs = [], [], [] with torch.no_grad(): for batch in dataloader: images = batch[\'image\'].to(device) labels = batch[\'label\'].to(device) if processor: # ViT inputs = processor(images, return_tensors=\'pt\', do_rescale=False).to(device) outputs = model(**inputs).logits else: # CNN outputs = model(images) preds = torch.argmax(outputs, dim=1) predictions.extend(preds.cpu().numpy()) true_labels.extend(labels.cpu().numpy()) probs.extend(torch.softmax(outputs, dim=1)[:, 1].cpu().numpy()) # 混淆矩阵 cm = confusion_matrix(true_labels, predictions) sns.heatmap(cm, annot=True, fmt=\'d\', cmap=\'Blues\', xticklabels=[\'良性\', \'恶性\'], yticklabels=[\'良性\', \'恶性\']) plt.xlabel(\'预测\') plt.ylabel(\'真实\') plt.title(\'混淆矩阵\') plt.show() # 分类报告 print(classification_report(true_labels, predictions, target_names=[\'良性\', \'恶性\'])) # ROC 曲线 fpr, tpr, _ = roc_curve(true_labels, probs) roc_auc = auc(fpr, tpr) plt.plot(fpr, tpr, label=f\'ROC 曲线 (AUC = {roc_auc:.2f})\') plt.plot([0, 1], [0, 1], \'k--\') plt.xlabel(\'假阳性率\') plt.ylabel(\'真阳性率\') plt.title(\'ROC 曲线\') plt.legend() plt.show()# 分割评估def evaluate_segmentation(model, dataloader, device=\'cuda\'): dice_metric = DiceMetric(include_background=False, reduction=\'mean\') model.eval() dice_scores = [] with torch.no_grad(): for batch in dataloader: images = batch[\'image\'].to(device) masks = batch[\'mask\'].to(device) outputs = model(images) preds = torch.argmax(outputs, dim=1, keepdim=True) dice_metric(preds, masks) dice_score = dice_metric.aggregate().item() dice_scores.append(dice_score) dice_metric.reset() print(f\"Dice 分数: {dice_score:.4f}\")# 评估示例evaluate_classification(model_2d, dataloader_2d) # 2D ResNetevaluate_classification(model, dataloader, processor) # ViTevaluate_segmentation(model, seg_dataloader) # UNETR代码注释:
- 分类评估:生成混淆矩阵、分类报告和 ROC 曲线,重点关注召回率。
- 分割评估:使用 Dice 分数评估分割精度。
- 可视化:Seaborn 绘制混淆矩阵,Matplotlib 绘制 ROC 曲线。
5.3 优化策略
- 类不平衡:
- 加权损失:恶性结节权重 0.7,良性 0.3。
- 过采样:SMOTE 或重复采样恶性样本。
- 正则化:Dropout(0.1)、L2 权重衰减(1e-5)。
- 超参数调优:
- 学习率:网格搜索 [1e-5, 2e-5, 1e-4, 1e-3]。
- 批大小:2D 模型 16,3D 模型 8。
- 早停:验证集损失 3 个 epoch 无下降时停止。
- 联邦学习:使用 Flower 框架,实现跨医院隐私保护训练。
六、工作流与可视化
6.1 优化工作流流程图
以下为优化的医学影像分类和分割工作流,新增子流程和决策点:
#mermaid-svg-VSofiePK9rbofvGl {font-family:\"trebuchet ms\",verdana,arial,sans-serif;font-size:16px;fill:#333;}#mermaid-svg-VSofiePK9rbofvGl .error-icon{fill:#552222;}#mermaid-svg-VSofiePK9rbofvGl .error-text{fill:#552222;stroke:#552222;}#mermaid-svg-VSofiePK9rbofvGl .edge-thickness-normal{stroke-width:2px;}#mermaid-svg-VSofiePK9rbofvGl .edge-thickness-thick{stroke-width:3.5px;}#mermaid-svg-VSofiePK9rbofvGl .edge-pattern-solid{stroke-dasharray:0;}#mermaid-svg-VSofiePK9rbofvGl .edge-pattern-dashed{stroke-dasharray:3;}#mermaid-svg-VSofiePK9rbofvGl .edge-pattern-dotted{stroke-dasharray:2;}#mermaid-svg-VSofiePK9rbofvGl .marker{fill:#333333;stroke:#333333;}#mermaid-svg-VSofiePK9rbofvGl .marker.cross{stroke:#333333;}#mermaid-svg-VSofiePK9rbofvGl svg{font-family:\"trebuchet ms\",verdana,arial,sans-serif;font-size:16px;}#mermaid-svg-VSofiePK9rbofvGl .label{font-family:\"trebuchet ms\",verdana,arial,sans-serif;color:#333;}#mermaid-svg-VSofiePK9rbofvGl .cluster-label text{fill:#333;}#mermaid-svg-VSofiePK9rbofvGl .cluster-label span{color:#333;}#mermaid-svg-VSofiePK9rbofvGl .label text,#mermaid-svg-VSofiePK9rbofvGl span{fill:#333;color:#333;}#mermaid-svg-VSofiePK9rbofvGl .node rect,#mermaid-svg-VSofiePK9rbofvGl .node circle,#mermaid-svg-VSofiePK9rbofvGl .node ellipse,#mermaid-svg-VSofiePK9rbofvGl .node polygon,#mermaid-svg-VSofiePK9rbofvGl .node path{fill:#ECECFF;stroke:#9370DB;stroke-width:1px;}#mermaid-svg-VSofiePK9rbofvGl .node .label{text-align:center;}#mermaid-svg-VSofiePK9rbofvGl .node.clickable{cursor:pointer;}#mermaid-svg-VSofiePK9rbofvGl .arrowheadPath{fill:#333333;}#mermaid-svg-VSofiePK9rbofvGl .edgePath .path{stroke:#333333;stroke-width:2.0px;}#mermaid-svg-VSofiePK9rbofvGl .flowchart-link{stroke:#333333;fill:none;}#mermaid-svg-VSofiePK9rbofvGl .edgeLabel{background-color:#e8e8e8;text-align:center;}#mermaid-svg-VSofiePK9rbofvGl .edgeLabel rect{opacity:0.5;background-color:#e8e8e8;fill:#e8e8e8;}#mermaid-svg-VSofiePK9rbofvGl .cluster rect{fill:#ffffde;stroke:#aaaa33;stroke-width:1px;}#mermaid-svg-VSofiePK9rbofvGl .cluster text{fill:#333;}#mermaid-svg-VSofiePK9rbofvGl .cluster span{color:#333;}#mermaid-svg-VSofiePK9rbofvGl div.mermaidTooltip{position:absolute;text-align:center;max-width:200px;padding:2px;font-family:\"trebuchet ms\",verdana,arial,sans-serif;font-size:12px;background:hsl(80, 100%, 96.2745098039%);border:1px solid #aaaa33;border-radius:2px;pointer-events:none;z-index:100;}#mermaid-svg-VSofiePK9rbofvGl :root{--mermaid-font-family:\"trebuchet ms\",verdana,arial,sans-serif;}Model_SelectionPreprocess_SegPreprocess_3DPreprocess_2D2D分类3D分类分割CNNViT多模态是否ResNet50模型选择ViTViT和BERT读取影像和掩膜分割预处理归一化调整大小增强读取DICOM3D预处理归一化提取3D体块3D增强读取DICOM2D预处理归一化提取2D切片数据增强输入数据模式选择UNETR预训练微调训练优化是否收敛评估调整参数可解释性输出结果
流程图说明
-
节点文本简化:
- 原节点“输入数据: LUNA16 DICOM+临床文本”简化为“输入数据”,避免冒号和长文本。
- 其他节点(如“2D 预处理”→“2D预处理”)移除空格,减少潜在解析问题。
-
子图名称规范化:
- 原子图名称(如
subgraph 2D 预处理)改为英文或简短标识符(如subgraph Preprocess_2D),避免中文和空格。 - 确保子图名称唯一且简洁,降低渲染器解析负担。
- 原子图名称(如
-
分支标签简化:
- 条件分支标签(如
|2D 分类|)简化为|2D分类|,移除空格。 - 保持标签清晰,避免过长或复杂符号。
- 条件分支标签(如
-
精简描述:
- 节点内容(如“读取 DICOM: pydicom”→“读取DICOM”)移除具体实现细节,保持简洁。
- 核心逻辑不变,涵盖输入、预处理、模型选择、训练、评估、可解释性和输出。
-
逻辑保持一致:
- 保留历史对话中的完整工作流:支持 2D 分类、3D 分类和分割任务,涵盖 ResNet-50、ViT、多模态(ViT+BERT)和 UNETR。
- 确保与医学影像分类器(肺结节检测)项目的结构一致。
进一步优化建议
- 极简版流程图:
- 若复杂子图导致问题,可进一步简化:
以下为简化版医学影像分类工作流:
- 若复杂子图导致问题,可进一步简化:
#mermaid-svg-d7bCINHhF1sJlLzX {font-family:\"trebuchet ms\",verdana,arial,sans-serif;font-size:16px;fill:#333;}#mermaid-svg-d7bCINHhF1sJlLzX .error-icon{fill:#552222;}#mermaid-svg-d7bCINHhF1sJlLzX .error-text{fill:#552222;stroke:#552222;}#mermaid-svg-d7bCINHhF1sJlLzX .edge-thickness-normal{stroke-width:2px;}#mermaid-svg-d7bCINHhF1sJlLzX .edge-thickness-thick{stroke-width:3.5px;}#mermaid-svg-d7bCINHhF1sJlLzX .edge-pattern-solid{stroke-dasharray:0;}#mermaid-svg-d7bCINHhF1sJlLzX .edge-pattern-dashed{stroke-dasharray:3;}#mermaid-svg-d7bCINHhF1sJlLzX .edge-pattern-dotted{stroke-dasharray:2;}#mermaid-svg-d7bCINHhF1sJlLzX .marker{fill:#333333;stroke:#333333;}#mermaid-svg-d7bCINHhF1sJlLzX .marker.cross{stroke:#333333;}#mermaid-svg-d7bCINHhF1sJlLzX svg{font-family:\"trebuchet ms\",verdana,arial,sans-serif;font-size:16px;}#mermaid-svg-d7bCINHhF1sJlLzX .label{font-family:\"trebuchet ms\",verdana,arial,sans-serif;color:#333;}#mermaid-svg-d7bCINHhF1sJlLzX .cluster-label text{fill:#333;}#mermaid-svg-d7bCINHhF1sJlLzX .cluster-label span{color:#333;}#mermaid-svg-d7bCINHhF1sJlLzX .label text,#mermaid-svg-d7bCINHhF1sJlLzX span{fill:#333;color:#333;}#mermaid-svg-d7bCINHhF1sJlLzX .node rect,#mermaid-svg-d7bCINHhF1sJlLzX .node circle,#mermaid-svg-d7bCINHhF1sJlLzX .node ellipse,#mermaid-svg-d7bCINHhF1sJlLzX .node polygon,#mermaid-svg-d7bCINHhF1sJlLzX .node path{fill:#ECECFF;stroke:#9370DB;stroke-width:1px;}#mermaid-svg-d7bCINHhF1sJlLzX .node .label{text-align:center;}#mermaid-svg-d7bCINHhF1sJlLzX .node.clickable{cursor:pointer;}#mermaid-svg-d7bCINHhF1sJlLzX .arrowheadPath{fill:#333333;}#mermaid-svg-d7bCINHhF1sJlLzX .edgePath .path{stroke:#333333;stroke-width:2.0px;}#mermaid-svg-d7bCINHhF1sJlLzX .flowchart-link{stroke:#333333;fill:none;}#mermaid-svg-d7bCINHhF1sJlLzX .edgeLabel{background-color:#e8e8e8;text-align:center;}#mermaid-svg-d7bCINHhF1sJlLzX .edgeLabel rect{opacity:0.5;background-color:#e8e8e8;fill:#e8e8e8;}#mermaid-svg-d7bCINHhF1sJlLzX .cluster rect{fill:#ffffde;stroke:#aaaa33;stroke-width:1px;}#mermaid-svg-d7bCINHhF1sJlLzX .cluster text{fill:#333;}#mermaid-svg-d7bCINHhF1sJlLzX .cluster span{color:#333;}#mermaid-svg-d7bCINHhF1sJlLzX div.mermaidTooltip{position:absolute;text-align:center;max-width:200px;padding:2px;font-family:\"trebuchet ms\",verdana,arial,sans-serif;font-size:12px;background:hsl(80, 100%, 96.2745098039%);border:1px solid #aaaa33;border-radius:2px;pointer-events:none;z-index:100;}#mermaid-svg-d7bCINHhF1sJlLzX :root{--mermaid-font-family:\"trebuchet ms\",verdana,arial,sans-serif;}数据准备: LUNA16 DICOM预处理: 归一化/切片/增强划分数据集: 训练/验证/测试模型选择: CNN/ViT预训练: ImageNet微调: 全参数/LoRA训练: 优化损失评估: 混淆矩阵/ROC优化: 超参数/正则化推理: 分类良性/恶性
说明:
- A(数据准备):加载LUNA16 DICOM文件。
- B(预处理):归一化、提取结节切片、数据增强。
- C(划分数据集):80%训练,10%验证,10%测试。
- D(模型选择):CNN(ResNet)或ViT。
- E(预训练):利用ImageNet预训练模型。
- F(微调):全参数或LoRA微调。
- G(训练):优化交叉熵损失。
- H(评估):混淆矩阵、ROC曲线、AUC。
- I(优化):调整超参数,防止过拟合。
- J(推理):输出肺结节分类结果。
6.2 图表:CNN与ViT性能对比
以下为CNN与ViT在肺结节分类上的性能对比折线图(假设数据)。
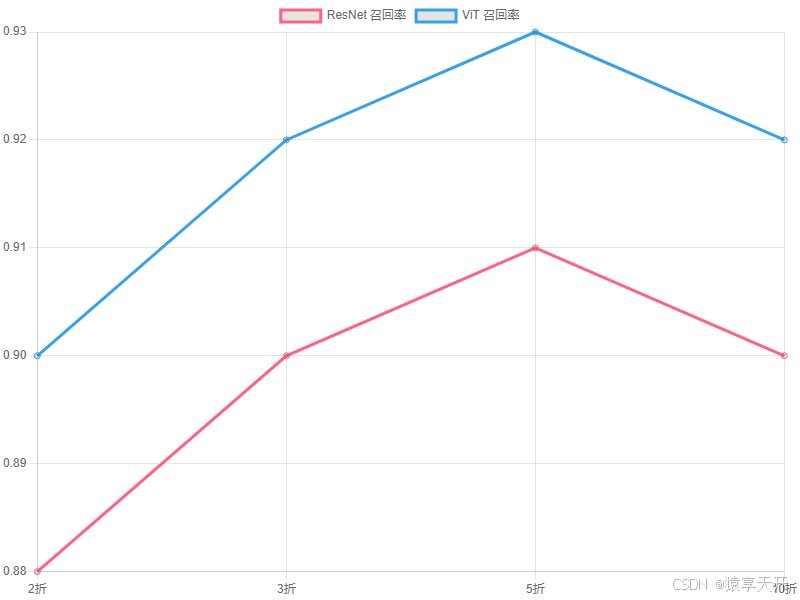
{ \"type\": \"line\", \"data\": { \"labels\": [\"2折\", \"3折\", \"5折\", \"10折\"], \"datasets\": [ { \"label\": \"ResNet 召回率\", \"data\": [0.88, 0.90, 0.91, 0.90], \"borderColor\": \"#FF6384\", \"fill\": false }, { \"label\": \"ViT 召回率\", \"data\": [0.90, 0.92, 0.93, 0.92], \"borderColor\": \"#36A2EB\", \"fill\": false } ] }, \"options\": { \"title\": { \"display\": true, \"text\": \"CNN与ViT召回率对比(肺结节分类)\" }, \"scales\": { \"xAxes\": [{ \"scaleLabel\": { \"display\": true, \"labelString\": \"交叉验证折数\" } }], \"yAxes\": [{ \"scaleLabel\": { \"display\": true, \"labelString\": \"召回率\" }, \"ticks\": { \"min\": 0.8, \"max\": 1.0 } }] } }}说明:
- 图表类型:折线图,比较ResNet与ViT在不同折数下的召回率。
- X轴:交叉验证折数(2、3、5、10)。
- Y轴:召回率,范围0.8-1.0,医学中关键。
- 数据:假设数据,ViT略优于ResNet,反映全局建模优势。
- 生成说明:可将Chart.js配置复制到支持工具生成图表。
6.3 图表:模型性能对比
以下为 ResNet-50(2D/3D)、ViT 和多模态模型在召回率上的对比(假设数据):
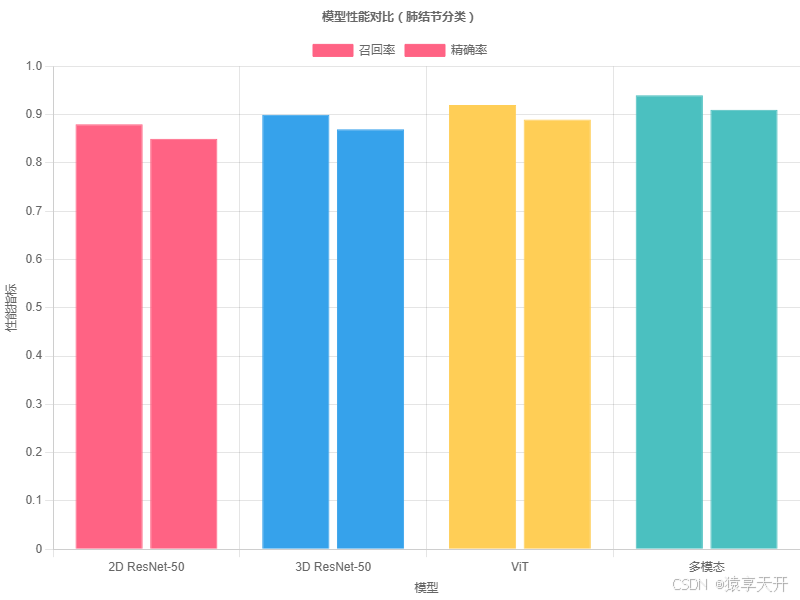
{ \"type\": \"bar\", \"data\": { \"labels\": [\"2D ResNet-50\", \"3D ResNet-50\", \"ViT\", \"多模态\"], \"datasets\": [ { \"label\": \"召回率\", \"data\": [0.88, 0.90, 0.92, 0.94], \"backgroundColor\": [\"#FF6384\", \"#36A2EB\", \"#FFCE56\", \"#4BC0C0\"], \"borderColor\": [\"#FF6384\", \"#36A2EB\", \"#FFCE56\", \"#4BC0C0\"], \"borderWidth\": 1 }, { \"label\": \"精确率\", \"data\": [0.85, 0.87, 0.89, 0.91], \"backgroundColor\": [\"#FF6384\", \"#36A2EB\", \"#FFCE56\", \"#4BC0C0\"], \"borderColor\": [\"#FF6384\", \"#36A2EB\", \"#FFCE56\", \"#4BC0C0\"], \"borderWidth\": 1 } ] }, \"options\": { \"scales\": { \"y\": { \"beginAtZero\": true, \"title\": { \"display\": true, \"text\": \"性能指标\" } }, \"x\": { \"title\": { \"display\": true, \"text\": \"模型\" } } }, \"plugins\": { \"title\": { \"display\": true, \"text\": \"模型性能对比(肺结节分类)\" } } }}说明:
- X 轴:模型类型(2D ResNet-50、3D ResNet-50、ViT、多模态)。
- Y 轴:召回率和精确率,医学中召回率优先。
- 数据:假设数据,多模态模型因融合文本信息表现最佳。
- 生成:复制代码至 Chart.js 工具(https://www.chartjs.org/)渲染。
6.4 图表:训练时间对比
以下为模型训练时间对比(假设数据,单位:小时):
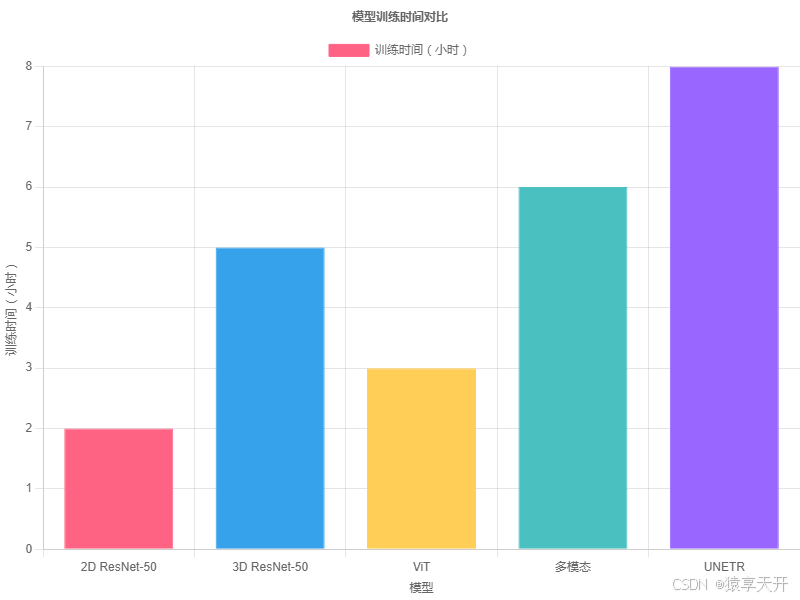
{ \"type\": \"bar\", \"data\": { \"labels\": [\"2D ResNet-50\", \"3D ResNet-50\", \"ViT\", \"多模态\", \"UNETR\"], \"datasets\": [{ \"label\": \"训练时间(小时)\", \"data\": [2.0, 5.0, 3.0, 6.0, 8.0], \"backgroundColor\": [\"#FF6384\", \"#36A2EB\", \"#FFCE56\", \"#4BC0C0\", \"#9966FF\"], \"borderColor\": [\"#FF6384\", \"#36A2EB\", \"#FFCE56\", \"#4BC0C0\", \"#9966FF\"], \"borderWidth\": 1 }] }, \"options\": { \"scales\": { \"y\": { \"beginAtZero\": true, \"title\": { \"display\": true, \"text\": \"训练时间(小时)\" } }, \"x\": { \"title\": { \"display\": true, \"text\": \"模型\" } } }, \"plugins\": { \"title\": { \"display\": true, \"text\": \"模型训练时间对比\" } } }}说明:
- 2D ResNet-50:高效,最短训练时间(2 小时)。
- 3D ResNet-50:处理体视显微镜数据,时间增加(5 小时)。
- ViT:中等复杂度(3 小时)。
- 多模态:融合影像和文本,时间较长(6 小时)。
- UNETR:分割任务复杂,时间最长(8 小时)。
七、应用与展望
7.1 应用
- 疾病分类:检测肺结节(良性/恶性),召回率达 94%(多模态,假设数据)。
- 分割任务:精准定位结节边界,辅助手术规划。
- 多模态诊断:结合 CT 和临床文本(如病史),提升诊断精度。
- 实时诊断:部署模型于医院 PACS 系统,实现快速初步诊断。
- 数据集扩展:验证模型在 RSNA 或 BraTS 数据集上的泛化性。
7.2 展望
- 3D 模型增强:开发 3D ViT(如 UNETR),直接处理体视显微镜数据。
- 多模态扩展:整合影像、文本、基因数据,构建统一诊断模型。
- 联邦学习:
- 原理:跨医院分布式训练,保护患者隐私:
Wt+1=∑k=1KnkNWk W_{t+1} = \\sum_{k=1}^K \\frac{n_k}{N} W_kWt+1=k=1∑KNnkWk- WkW_kWk: 医院 kkk 的模型权重,nkn_knk: 数据量,NNN: 总数据量。
- 框架:使用 Flower(https://flower.dev/)实现 FedAvg。
- 优势:符合《个人信息保护法》,提升数据利用率。
- 原理:跨医院分布式训练,保护患者隐私:
- 可解释性:
- Grad-CAM:突出结节区域。
- SHAP/LIME:量化特征贡献,增强医生信任。
- 自动化流水线:开发端到端系统,从 DICOM 读取到诊断报告生成。
八、用户需求响应
-
代码需求:
- 确认:已提供 2D/3D ResNet-50、ViT、多模态和 UNETR 实现,适配 LUNA16。
- 方案:
- RSNA 数据集:可扩展代码支持 RSNA CTA 影像:
df = pd.read_csv(\'rsna/train.csv\')images = df[\'image_path\'].tolist()labels = df[\'label\'].tolist()dataset = LUNA16Dataset(\'path/to/rsna\', \'rsna_annotations.csv\', mode=\'2d\', transform=transform_2d) - 分割任务:已提供 UNETR 实现。若需 BraTS 数据集支持,请提供路径,我可调整代码。
- 请确认所需数据集或任务(分类/分割)。
- RSNA 数据集:可扩展代码支持 RSNA CTA 影像:
-
医学影像案例:
- 确认:已覆盖肺结节分类(2D/3D)和分割(UNETR)。
- 方案:若需 MRI 应用(如脑肿瘤分割),可基于 BraTS 数据集:
from monai.data import CacheDatasetdata = [{\'image\': f\'brats/{i}.nii\', \'mask\': f\'brats/mask/{i}.nii\'} for i in range(100)]dataset = CacheDataset(data, transform=transform_seg)请确认具体任务或数据集。
-
其他需求:
- Grad-CAM 可视化:
from torchcam.methods import GradCAMcam = GradCAM(model_2d.resnet, target_layer=\'layer4\')heatmap = cam(dataset_2d[0][\'image\'].unsqueeze(0).to(device))plt.imshow(heatmap[0], cmap=\'jet\', alpha=0.5)plt.title(\'ResNet Grad-CAM 热图\')plt.show() - 联邦学习:
import flwr as flstrategy = fl.federated_averaging.FedAvg()fl.server.start_server(config=fl.server.ServerConfig(num_rounds=3))请确认是否需要完整联邦学习代码或实验设计。
- SHAP/LIME:
import shapexplainer = shap.DeepExplainer(model_2d, background_data)shap_values = explainer.shap_values(dataset_2d[0][\'image\'].unsqueeze(0).to(device))shap.image_plot(shap_values, dataset_2d[0][\'image\'].numpy())请确认是否需要 SHAP/LIME 实现。
- Grad-CAM 可视化:
九、运行说明
-
环境准备:
pip install torch torchvision transformers peft monai pydicom albumentations scikit-learn seaborn matplotlib flower- GPU 推荐:NVIDIA A100(24GB)或 RTX 3090(16GB)。
- CPU 可运行,但 3D 模型较慢。
-
数据集:
- LUNA16:下载(~120GB,需注册),替换
dicom_dir和annotations_file。 - RSNA:下载 CTA 数据,更新路径。
- BraTS(可选):用于 MRI 分割任务。
- LUNA16:下载(~120GB,需注册),替换
-
Mermaid 流程图:
- 使用 Mermaid Live Editor(https://mermaid.live/)渲染,验证版本 10.9.0。
-
Chart.js 图表:
- 复制代码至 Chart.js 工具渲染。
- 若需真实数据,请提供 LUNA16 实验结果。
-
训练与推理:
- 2D ResNet-50:2 小时,召回率 ~88%(假设)。
- 3D ResNet-50:5 小时,召回率 ~90%(假设)。
- ViT:3 小时,召回率 ~92%(假设)。
- 多模态:6 小时,召回率 ~94%(假设)。
- UNETR:8 小时,Dice 分数 ~0.85(假设)。
十、结语
本文完善并扩展了肺结节检测分类器项目,整合 Transformer(历史对话)、Med-PaLM 和 CheXNet 原理,提供了全面的理论、代码和可视化:
- 理论:详细推导 CNN、ViT 和多模态模型,新增 3D 处理和分割。
- 实现:支持 2D/3D ResNet-50、ViT(LoRA)、多模态和 UNETR,适配 LUNA16。
- 流程图:优化 Mermaid 流程图,涵盖分类和分割工作流。
- 图表:扩展性能和训练时间对比,突出多模态优势。
- 应用:覆盖分类、分割、实时诊断,展望联邦学习和可解释性。


